BACTEC
MGITä Pozitif Kültürlerden
Mycobacterium Türlerinin
Oligo-FISH ve PNA-FISH Yöntemleriyle Tanımlanması*
Identification
of Mycobacterium Species from BACTEC MGIT
Positive Cultures with Oligo-FISH and PNA-FISH Methods
Gülay BÖREKÇİ1, Gönül ASLAN2, Esin AYDIN2, Mark J. FIANDACA3, Henrik STENDER3, Natuschka M. LEE4,
Yusuf ÖZKUL5, Gürol EMEKDAŞ2
1 Mersin Üniversitesi Sağlık Yüksekokulu, Mersin.
1 Mersin University School of Health Sciences, Mersin, Turkey.
2 Mersin Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Mersin.
2 Mersin University Faculty of Medicine, Department of Medical Microbiology, Mersin, Turkey.
3 AdvanDx Inc. Firması, Woburn, Massachusetts, ABD.
3 AdvanDx, Inc., Woburn, MA 01801, USA.
4 Münih Teknik Üniversitesi, Mikrobiyoloji Bölümü, Freising, Almanya.
4 Technische Universität München, Department of Microbiology, D-85354 Freising, Germany
5 Erciyes Üniversitesi Tıp Fakültesi, Tıbbi Genetik Anabilim Dalı, Kayseri.
5 Erciyes University Faculty of Medicine, Department of Medical Genetics, Kayseri, Turkey.
* Bu çalışma, Mersin Üniversitesi Bilimsel Araştırma ve Projeleri Birimi tarafından desteklenmiştir.
ÖZET
Mikobakterilerin hızlı ve doğru tanısı, günümüzde halen ciddi bir halk sağlığı sorunu olan tüberkülozun önlenmesi ve hastaların etkin tedavisi için önem taşımaktadır. Fluoresan in situ hibridizasyon (FISH) yöntemi, rRNA'yı hedef alan problar kullanılarak direkt klinik örnekler ve kültürlerden, karışık ortamdaki mikroorganizmaların birkaç saat içinde kesin ve doğru tanımlanmasına olanak sağlamaktadır. Bu çalışmada, BACTEC MGIT (Mycobacteria Growth Indicator Tube) pozitif kültürlerden mikobakteri türlerinin tanımlanması için iki farklı FISH (Oligo-FISH ve PNA-FISH) yönteminin konvansiyonel yöntemlerle karşılaştırılarak rutin laboratuvar kullanımındaki tanısal performansının değerlendirilmesi amaçlanmıştır. Çalışmaya, 60 MGIT (BD, ABD) pozitif ve 52 MGIT negatif kültür ile 10 farklı referans suş dahil edilmiştir. Oligo-FISH yönteminde, 16S rRNA'yı hedef alan oligonükleotid problar (Myc657: Mycobacterium subdivision, Eub338: Pozitif kontrol, NonEub: Negatif kontrol); PNA-FISH yönteminde ise 16S rRNA'yı hedef alan peptid nükleik asit problar (MTC: Mycobacterium tuberculosis kompleks, NTM: Tüberküloz dışı mikobakteri, BacUni: Pozitif kontrol) kullanılmıştır. Konvansiyonel yöntemler olarak, MGIT 960 kültür sisteminin yanı sıra Ehrlich-Ziehl-Neelsen boyama (ARB) ve Löwenstein-Jensen (LJ) besiyerinde kültür yöntemleri uygulanmıştır. Çalışmaya alınan MGIT pozitif 60 (44 balgam, 4 doku, 4 idrar, 3 bronkoalveoler lavaj, 3 BOS, 1 apse, 1 periton sıvısı) kültürden 29 (%48.3)'u ARB ile, 44 (%73.3)'ü LJ kültür yöntemi ile olmak üzere toplam 59'u pozitif sonuç vermiş; bunların 58 (%96.6)'i MTC, 1 (%1.7)'i ise NTM olarak tiplendirilmiştir. Oligo-FISH ile izolatların %95 (57/60)'i Mycobacterium spp. olarak saptanırken, 3 (%5)ü negatif bulunmuştur. PNA-FISH ile 54 (%91.5) izolat mikobakteri olarak saptanmış ve bu izolatlardan 53 (%90)'ü MTC, 1 (%1.7)'i NTM olarak tiplendirilmiştir. Oligo-FISH ile pozitif, PNA-FISH ile negatif sonuç veren beş izolatın tanımlanmasında ise, PNA-FISH yönteminde yapılan minör modifikasyon ile pozitif sonuç elde edilmiştir. Oligo- ve PNA-FISH yöntemlerinin, MGIT pozitif kültürlerden mikobakterileri tanımlama süresi konvansiyonel yöntemlere göre daha kısa olup (2-2.5 saat), her iki FISH yönteminin konvansiyonel yöntemlere göre, PNA-FISH yönteminin de Oligo-FISH'e göre daha pratik olduğu saptanmıştır. Oligo-FISH yönteminin duyarlılık, özgüllük, pozitif ve negatif prediktif değeri sırasıyla, %96.6, %100, %100 ve %96.4 olarak belirlenmiş; bu değerler PNA-FISH için sırasıyla, %91.5, %100, %100 ve %91.4 olarak bulunmuştur (p< 0.0001). Yöntemlerin uyumluluğu kappa istatistiksel analizi kullanılarak belirlenmiş; her iki FISH yöntemi arasında ve her ikisi ile konvansiyonel yöntemler arasında mükemmel uyumluluk saptanmıştır (: 0.964, 0.929, 0.964; p= 0.001). Çalışmada ayrıca, kullanılan oligonükleotid ve PNA probların kapsamı SILVA 102 16S rRNA veri tabanı kullanılarak kontrol edilmiştir. Probların veri tabanında yer alan mikobakterileri sırasıyla; Eub338 için %86.5, Myc657 için %41.7, BacUni için %84.2, MTC için %76.3 (sadece M.tuberculosis ve M.bovis için %100) ve NTM için %25.8 oranında kapsadığı belirlenmiştir. Sonuç olarak, rutin mikobakteriyoloji laboratuvarlarında, MGIT pozitif kültürlerden mikobakteri türlerinin, pahalı yöntemlere gereksinim olmaksızın, hızlı ve doğru tanısı için Oligo- ve PNA-FISH yöntemlerinin kullanılabileceği kanısına varılmıştır.
Anahtar sözcükler: Mikobakteri türleri; tanımlama; in situ hibridizasyon; PNA-FISH; Oligo-FISH; konvansiyonel yöntemler.
ABSTRACT
Rapid and accurate diagnosis of mycobacteria is very important in the prevention and effective treatment of tuberculosis which is still a serious public health problem. Fluorescence in situ hybridization (FISH) method using rRNA targeted probes allows for precise and accurate identification of mixed microorganisms from cultures and directly from clinical samples within a few hours without the need for culture methods. In this study it was aimed to compare the diagnostic performance of two different FISH methods (Oligo-FISH and PNA-FISH) with the conventional culture methods for the identification of Mycobacterium spp. grown in BACTEC MGIT (Mycobacteria Growth Indicator Tube) system. A total of 60 MGIT (BD, USA) positive, 52 MGIT negative samples and 10 different reference strains were included in the study. 16S rRNA targeted oligonucleotide probes (Myc657: Mycobacterium subdivision, Eub338: Positive control, NonEub: Negative control) were used for oligo-FISH, and 16S rRNA targeted peptide nucleotide probes (MTC: Mycobacterium tuberculosis complex, NTM: Non-tuberculosis Mycobacterium, BacUni: Positive control) for PNA-FISH. Ehrlich-Ziehl-Neelsen staining (ARB) and Löwenstein-Jensen (LJ) culture methods were performed as conventional methods as well as MGIT 960 culture system. Of MGIT positive 60 samples (44 sputum, 4 tissue, 4 urine, 3 bronchoalveolar lavage, 3 CSF, 1 abscess, 1 peritoneal fluid), 29 (48.3%) were found positive for ARB and 44 (73.3%) with LJ culture methods giving a total of 59 positive results. Fifty-eight (96.6%) of those isolates were identified as MTC, and one (1.7%) as NTM by conventional methods. By using Oligo-FISH, 95% (57/60) of the isolates were identified as Mycobacterium spp., while three samples (5%) yielded negative result. By using PNA-FISH, 54 (91.5%) isolates were identified as mycobacteria, of them 53 (90%) were typed as MTC and 1 (1.7%) as NTM. Five isolates that were found positive with Oligo-FISH, but negative with PNA-FISH, yielded positive result with PNA-FISH method performed with minor modifications. It was determined that both FISH methods are more rapid (approximately 2-2.5 hours) and practical than the conventional culture methods and also PNA-FISH was more practical than Oligo-FISH. The sensitivity, specificity, positive and negative predictive values of the probes used for Oligo-FISH, were 96.6%, 100%, 100% and 96.4%, respectively. Those values for the probes used for PNA-FISH, were 91.5%, 100%, 100% and 91.4%, respectively (p< 0.0001). The compatibility of the methods was calculated with kappa statistical analysis, assigning perfect concordances between Oligo- and PNA-FISH methods, as well as between conventional and both of the FISH methods (: 0.964, 0.929, 0.964; p= 0.001). The coverage of oligonucleotide and PNA probes was also checked by using 16S rRNA gene sequence database retrieved from the SILVA 102. It was determined that the rates of coverage were 86.5% for Eub338, 41.7% for Myc657, 84.2% for BacUni, 76.3% for MTC (100% for only M.tuberculosis and M.bovis) and 25.8% for NTM probes. In conclusion, Oligo- and PNA-FISH methods seem to be successful for rapid and accurate identification of Mycobacterium spp. from MGIT positive cultures in routine mycobacteriology laboratories without the need for expensive methods.
Key words: Mycobacterium species; in situ identification; PNA-FISH; Oligo-FISH; conventional culture methods.
Geliş Tarihi (Received): 14.03.2014 • Kabul Ediliş Tarihi (Accepted): 20.06.2014
GİRİŞ
Tüberküloz (TB), ülkemizde ve gelişmekte olan ülkelerde hala önemli bir halk sağlığı sorunudur. Dünya Sağlık Örgütü (DSÖ) 2012 verilerine göre, tüm dünyada 8.6 milyon TB olgusu bulunmakta ve 1.3 milyon insan TB nedeniyle ölmektedir1. Özellikle çok ilaca dirençli tüberküloz (ÇİD-TB) olgularının bazı ülkelerde yüksek oranlarda görüldüğü ve ölümlerin 170.000'inin ÇİD-TB hastaları olduğunu bildirmektedir1. Tüberkülozun önlenmesi ve tedavisi için mikobakterilerin hızlı ve doğru tanısı önem taşımaktadır.
Günümüzde mikobakterilerin tanımlanmasında konvansiyonel kültür yöntemleri rutin mikobakteriyoloji laboratuvarında hala altın standart olarak kabul edilmektedir. Ancak rutin uygulamada tanımlama, zaman alıcı ve emek-yoğun işlemler gerektirmektedir. Radyometrik olmayan MGIT (Mycobacteria Growth Indicator Tube, BD, ABD) gibi teknikler, tanı süresini haftalardan günlere kadar kısaltmakla birlikte yine de yeterli olmamakta ve tedavi gecikebilmektedir2,3,4. Son yıllarda, farklı moleküler teknikler mikobakterilerin doğrudan tanımlanması için hız, güvenilirlik ve performans açısından geleneksel yöntemlere göre daha fazla avantaj sunmaktadır. Bu yöntemler tanımlama süresini, haftalardan birkaç gün ve birkaç saate kadar kısaltmaktadır. Bunlar arasında en yaygın kullanılanlar, nükleik asit amplifikasyonuna dayalı yöntemler (polymerase chain reaction, PCR) ile hibridizasyon yöntemleridir5,6. PCR'ye dayalı yöntemlerin bazı avantajları (tanımlama limitinin düşük olması, kısa sürede tanı koyması vb.) olmasına rağmen bazı kısıtlılıkları da (inhibitör bileşiklere duyarlılık, kontaminasyon vb.) bulunmaktadır. Bununla birlikte PCR yönteminin en önemli dezavantajı diğer yöntemlere göre daha pahalı olmasıdır5,7,8. Floresan in situ hibridizasyon (FISH) yöntemi ise, 16S, 18S veya 23S rRNA'yı hedef alan floresan işaretli oligonükleotid problar kullanılarak, hedefin işaretlenmesi ve floresan mikroskobunda görüntülenmesi temeline dayanan ve kısa sürede sonuç veren moleküler bir yöntemdir. Bu yöntem ile kültürü yapılabilen veya yapılamayan mikroorganizmaların doğal ortamlarında izolasyon ve tanımlamaları kısa sürede yapılabilmektedir9,10. Çeşitli alanlarda pek çok farklı FISH tekniği uygulanmakla birlikte, oligonükleotid problarla yapılan Oligo-FISH ve peptid nükleik asit (PNA) problarla yapılan PNA-FISH, mikrobiyal ekoloji ve tıp alanlarında daha çok kullanılmaktadır10,11,12,13. Her iki FISH yönteminde tanımlama süresi kısa olup (yaklaşık 2-2.5 saat), bu yöntemlerin duyarlılığı, özgüllüğü, pozitif ve negatif prediktif değerleri yüksektir14,15,16,17,18.
Rutin mikobakteriyoloji laboratuvarlarında mikobakterilerin hızlı, kolay ve ucuz yöntemlerle tanımlanması oldukça önemlidir. Bu amaçla daha önceki çalışmalarda farklı FISH probları (oligonükleotid, PNA) geliştirilmiş ve her iki yöntem için yüksek duyarlılık ve özgüllük bildirilmiştir13,16,19,20,21. Bununla birlikte her iki yöntemin rutin mikobakteriyoloji laboratuvarındaki yararları, dezavantajları ve sınırlılıkları bilinmemektedir. Bu çalışmada, MGIT pozitif kültürlerden mikobakterilerin hızlı tanımlanmasında, oligonükleotid ve PNA probları kullanarak Oligo- ve PNA-FISH yöntemlerinin karşılaştırılması ve rutin mikobakteriyoloji laboratuvarında uygulanabilirliğinin değerlendirilmesi amaçlanmıştır.
GEREÇ ve YÖNTEM
Klinik Örnekler ve Referans Suşlar
Çalışmaya, Mersin Üniversitesi Tıp Fakültesi, Araştırma ve Uygulama Hastanesi, Mikobakteriyoloji Laboratuvarına TB tanısı için gönderilen çeşitli klinik örneklerden (44 balgam, 4 doku, 4 idrar, 3 bronkoalveoler lavaj, 3 BOS, 1 apse, 1 periton sıvısı) BACTEC MGIT 960 (Becton Dickinson Microbiology Systems, ABD) sisteminde pozitif sinyal veren 60 örnek ile negatif 52 örnek dahil edildi.
Çalışmada, Acinetobacter baumannii RSHMB, Aeromonas hydrophila ATCC 7966, Bacillus cereus ATCC 11778, Bacillus subtilis ATCC 6633, Enterococcus faecalis ATCC 2921, Escherichia coli ATCC 25922, Klebsiella pneumoniae ATCC 1031, Lactococcus garviae ATCC 49156, Mycobacterium abscessus ATCC 19977, Mycobacterium avium ATCC 15769, Mycobacterium aurum ATCC 23366, Mycobacterium fortuitum subsp. fortuitium ATCC 6841, Mycobacterium gordonae ATCC 14470, Mycobacterium kansasii ATCC 12478, Mycobacterium scrofulaceum ATCC 19981, Mycobacterium senegalense TMC 806, Mycobacterium tuberculosis H37RV, Mycobacterium vaccae TMC 1526, Pseudomonas aeruginosa ATCC 27853, Staphylococcus aureus ATCC 25923, Streptococcus pneumoniae ATCC 700669 referans suşları pozitif ve negatif kontrol olarak kullanıldı. Referans mikobakteri suşları Middlebrook 7H9 sıvı besiyerinde (BBL, Becton Dickinson Company, ABD) 37°C'de 10 gün, diğer referans suşlar ise nutrient sıvı besiyerinde (Merck, Almanya) 37°C'de 24 saat inkübe edildi. Mikobakteri ve diğer referans suşlarının kültürlerinden aşağıda belirtilen iki farklı fiksasyon (Oligo ve PNA-FISH) protokolüne göre preparat hazırlandı ve iki farklı FISH yöntemi uygulandı.
Problar
Çalışmada kullanılan oligonükleotid ve PNA probların özellikleri Tablo I'de gösterildi14,19,22,23. FITC/CY3 işaretli 16S rRNA'yı hedef alan oligonükleotid problar Microsynth (İsviçre) firmasından; PNA problar ise AdvanDx (Woburn, ABD) firmasından temin edildi. Çalışmada kullanılan probların kapsamı SILVA 16S rRNA (102 versiyon) veri tabanı kullanılarak belirlendi.
Bakterilerin Fiksasyonu
Bu amaçla, Amann24 tarafından önerilen ve bazı araştırıcılar20,25 tarafından modifiye edilen yöntem uygulandı. Gram-negatif referans suşlar ile MGIT pozitif ve negatif örneklerin fiksasyonunda %4 paraformaldehid (PFA) kullanıldı. Her kültürden yaklaşık 200 µl alınıp, 600 µl fiksasyon tamponu (%4 PFA) (1:3 v/v) ile karıştırıldı ve 0-4°C'de 10 dakika inkübe edildi. Daha sonra 8000 rpm'de santrifüj edildi ve iki kez 1xPBS (phosphate buffer saline) ile yıkandı. PFA ile fikse edilen bakteri hücreleri, etanol/1xPBS (1/1 v/v) ile muamele edildi ve kullanılıncaya kadar -20°C'de karanlıkta saklandı. Gram-pozitif referans suşların fiksasyonu ise etanol ile yapıldı. Bakteri kültürleri eşit hacimde (1/1 v/v) soğuk etanol ile karıştırıldıktan sonra fikse edilen örnekler kullanılıncaya kadar -20°C'de karanlıkta saklandı.
PNA-FISH yönteminde gram-negatif ve gram-pozitif referans suşlar ile MGIT pozitif ve negatif kültürlerin fiksasyonu için üretici firmanın (AdvanDx Inc, ABD) önerisi doğrultusunda aynı protokol kullanıldı. Bir damla fiksasyon solüsyonu teflon lam üzerine damlatılarak, üzerine her kültürden 10 µl eklendi ve hafifçe karıştırıldı. Daha sonra preparatlar 55°C'de 20 dakika bekletilip, %96'lık etanolde 5-10 dakika tutulduktan sonra havada kurumaya bırakıldı.
FISH Yöntemleri
Oligo-FISH yöntemi Amann24 tarafından uygulanan yönteme göre yapıldı. Fikse edilmiş örneklerden 5 µl alınarak teflon lam üzerine konuldu; preparat 45oC'de 20 dakika kurumaya bırakıldı. Daha sonra sırasıyla %50, %80 ve %100 etanolde 3'er dakika bekletildi. Her bir kuyucuğa 1 µl prob (50 ng/µl FITC işaretli prob; 30/50 ng/µl Cy3 işaretli prob) ve 9 µl hibridizasyon tamponu [%20 formamid, 5 M NaCI, 1 M Tris %10 SDS (sodyum dodesil sülfat), H2O] konularak, 46oC'de 1.5 saat hibridizasyona bırakıldı. Daha sonra preparat yıkama tamponu (5 M NaCI, 1 M Tris, 10% SDS, H2O) ile 46oC'de 15-20 dakika yıkandı. Sonrasında, %0.001'lik 4',6-diamidino-2-phenylindole (DAPI) ile 7-10 dakika boyandı ve havada kurutulan preparatın her bir kuyucuğuna citifluor damlatılarak floresan mikroskopta (Olympus BX51) DAPI, FITC ve CY3 uyumlu filtreler ile software program (Olympus DP-BSW) kullanılarak incelendi.
PNA-FISH yöntemi ise üretici firmanın (AdvanDx Inc, ABD) önerdiği protokole göre uygulandı. Daha önce her prob için ayrı hazırlanan teflon lam kaplı preparatların üzerindeki kuyucuğa hibridizasyon tamponu [%10 (wt/vol) dekstran sülfat, 10 mM NaCl, %30 (vol/vol) formamid, %0.1 (wt/vol) sodyum pirofosfat, %0.2 (wt/vol) polivinil-pirolidon, %0.2 (wt/vol) Ficoll, 5 mM disodyum EDTA, %0.1 (vol/vol) Triton X-100 ve 50 mM Tris-HCl (pH 8.0)] içinde süspansiyon hale getirilen MTC ve NTM problardan (100 nM) birer damla damlatılarak üzeri lamel ile kapatıldı ve 55oC'de 1.5 saat hibridizasyona bırakıldı. Daha sonra preparatların üzerindeki lamel kaldırılıp, önceden ısıtılmış yıkama solüsyonu (AdvanDx wash solution; Tris-buffered saline) içinde 55oC'de 30 dakika bekletildi; havada kurutulup, üzerine bir damla mounting media (AdvanDx) damlatılarak yukarıda belirtildiği gibi floresan mikroskopta incelendi.
Çalışmamızda Oligo- ve PNA-FISH protokolünde minör modifikasyonlar uygulandı. Oligo-FISH yönteminde; referans mikobakteri suşları 20 ile 120 ng/µl arasında değişen aralıklarda prob konsantrasyonu ve farklı hibridizasyon süreleri (1.5 ve 16 saat) uygulanarak optimize edilmeye çalışıldı. Ayrıca Oligo-FISH yöntemiyle pozitif, ancak PNA-FISH yöntemiyle negatif sonuç veren bazı MGIT pozitif örneklerin fiksasyon protokolü değiştirildi ve bu örneklerin PNA-FISH ile tanımlanmasında PNA-FISH fiksasyon protokolü yerine Oligo-FISH fiksasyon protokolü uygulandı.
Mikobakterilerin Konvansiyonel Yöntemlerle Tanımlanması
Mikobakterilerin tanımlanmasında konvansiyonel yöntemler olarak; Ehrlich-Ziehl-Neelsen boyama (ARB), Löwenstein-Jensen (LJ) besiyeri ve MGIT 960 sistemi kullanıldı. MGIT pozitif kültürler ARB boyama ile doğrulandıktan sonra, tanımlama için PNB (para-nitro benzoik asit) besiyerinde üreme, nitrat, katalaz ve niasin gibi biyokimyasal testler yapılarak MTC (M.tuberculosis kompleks) ve NTM (Tüberküloz dışı mikobakteri) ayrımı yapıldı ve MTC olarak tanımlanan izolatlara anti-tüberküloz ilaç duyarlılık testi uygulandı2,26,27.
BULGULAR
Konvansiyonel Yöntem Sonuçları
Çalışmaya alınan 60 MGIT pozitif kültürden 59 (%98.3)'u konvansiyonel yöntemlerle Mycobacterium spp. olarak tanımlanmış; bir örnek ise konvansiyonel yöntemlerle negatif sonuç vermiştir. Bu 59 izolattan 58 (%96.6)'i M.tuberculosis kompleks (MTC), 1 (%1.7)'i ise tüberküloz dışı mikobakteri (NTM) olarak tiplendirilmiştir. Örneklerin %48.3 (29/60)'ü ARB, %73.3 (44/60)'ü LJ kültür yöntemiyle pozitif bulunmuştur (Tablo II). Direnç testi yapılan 50 izolattan 33 (%55)'ünün etambutol, rifampisin, streptomisin ve izoniazide duyarlı, 17 (%28.3)'sinin ise bunlardan en az birine karşı dirençli olduğu saptanmıştır (Tablo II).
MGIT Pozitif Kültürlerin FISH Sonuçları
Oligo-FISH ile 60 izolatın 57 (%95)'si Mycobacterium spp. olarak saptanırken, 3 (%5)'ü negatif olarak belirlenmiştir. PNA-FISH ile 54 izolat (%91.5) mikobakteri olarak saptanmış ve bu izolatlardan 53 (%90)'ü MTC, 1 (%1.7)'i NTM olarak tiplendirilmiştir. Oligo-FISH ile pozitif, PNA-FISH ile negatif sonuç veren sadece 5 izolatın tanımlanmasında yukarıda bahsedilen minör modifikasyon uygulanmış ve pozitif sonuç elde edilmiştir.
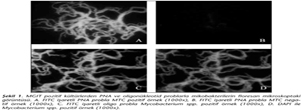
FISH yöntemi, ARB ve LJ kültür sonuçları ile karşılaştırıldığında; kültürle negatif olarak değerlendirilen 16 örnekten 14'ü; ARB ile negatif olan 31 örnekten 29'u FISH yöntemi ile pozitif bulunmuştur (Tablo II). Ayrıca MGIT sisteminde negatif olan 52 kültürün hiçbirinde LJ kültür ve FISH yöntemiyle mikobakteri saptanmamıştır. Floresan mikroskopta MGIT pozitif kültürlerin PNA- ve Oligo-FISH yöntemiyle incelenmesinde; her ikisinde de prob sinyal yoğunluğunun benzer olduğu (Şekil 1), ancak PNA-FISH yönteminde özgül olmayan bağlanmaların daha az olduğu görülmüştür.
Referans Suşların Oligo- ve PNA-FISH Sonuçları
Referans mikobakteri suşlarının Oligo- ve PNA-FISH sonuçları Tablo III'te verilmiştir. Oligonükleotid problar (Eub338, NonEub, Myc657) ile PNA probların (BacUni, MTC, NTM) özgüllüğü referans mikobakteri ve diğer gram-pozitif ve gram-negatif bakteri şuşları ile test edilmiştir. M.fortuitum hariç, çalışmada kullanılan tüm bakteri ve mikobakteri referans suşları bakterileri hedef alan genel problar (BacUni and Eub338; pozitif kontroller) ile pozitif reaksiyon vermiştir. Mycobacterium için özgül problar (Myc657, MTC, NTM) diğer referans suşlarla negatif reaksiyon verirken, M.fortuitum dışındaki referans mikobakteri suşları pozitif reaksiyon vermiştir. Her iki FISH çalışmasında da M.fortuitum'un otofloresan özellik gösterdiği belirlenmiştir. Oligo-FISH yönteminde NonEub prob (negatif kontrol) ise çoğu mikobakteri (M.abcessus, M.gordonae hariç) ve tüm diğer bakteri referans suşları ile negatif reaksiyon verirken, M.abcessus ve M.gordonae pozitif reaksiyon (özgül olmayan bağlanma) vermiştir. PNA-FISH yönteminde negatif prob mevcut olmadığı için bu bakterilerle ilgili reaksiyon değerlendirilememiştir. Bu suşların NonEub probu ile özgül olmayan bağlanmasını önlemek için Oligo-FISH yönteminde farklı prob konsantrasyonları (20 ng, 30 ng, 50 ng, 60 ng, 90 ng, 100 ng, 120 ng) ve hibridizasyon süreleri (1.5 ve 16 saat) uygulanmıştır. M.abcessus 30 ng ve M.gordonae 30 ng, 100 ng ve 120 ng prob konsantrasyonu ve 1.5 saatlik hibridizasyon süresinde özgül olmayan bağlanmanın olmadığı görülmüştür.
Her iki FISH yönteminde de bazı referans mikobakteri suşlarında farklı floresan sinyal yoğunlukları gözlemlenmiştir. M.tuberculosis, M.abcessus, M.vaccae ve M.aurum daha güçlü sinyal verirken, M.gordonae, M.senegalense ve M.scrofulaceum'un aynı preparatta iki farklı sinyal (kord formasyonu içinde zayıf, bu oluşumların dışında daha güçlü) verdikleri görülmüştür.
Probların SILVA 16S rRNA Veri Tabanındaki Mikobakterilerle Karşılaştırılması
Çalışmada kullanılan oligonükleotid ve PNA problar SILVA 102 [743 uzun diziler (> 1400 nükleotid), 5216 uzun ve kısa diziler (< 1400 nükleotid)] 16S rRNA veri tabanındaki mikobakterilerle karşılaştırılmış ve probların veri tabanında yer alan mikobakterileri sırasıyla Eub338 için %86.5, Myc657 için %41.7, BacUni için %84.2, MTC için %76.3 (sadece M.tuberculosis ve M.bovis için %100) ve NTM için %25.8 kapsadığı belirlenmiştir.
Oligo- ve PNA-FISH ile Konvansiyonel Kültür Yöntemlerinin Karşılaştırılması
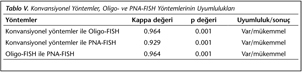
MGIT pozitif kültürlerden mikobakterilerin tanımlanmasında Oligo- ve PNA-FISH yöntemlerinin tanımlama süresi konvansiyonel yöntemlere göre daha kısa olup (2-2.5 saat), her iki FISH yönteminin konvansiyonel yöntemlere göre, PNA-FISH yönteminin de Oligo-FISH'e göre daha pratik olduğu saptanmıştır. Her iki yöntemde de konvansiyonel yöntemler altın standart kabul edilerek yöntemlerin duyarlılık, özgüllük, pozitif ve negatif prediktif değerleri hesaplanmıştır (Tablo IV). Ayrıca, konvansiyonel yöntemler, Oligo-FISH ve PNA-FISH yöntemleri arasında mükemmel uyum saptanmıştır (Tablo V).
TARTIŞMA
Mikobakterilerin tanımlanmasında konvansiyonel kültür yöntemleri hala altın standart olarak kabul edilmekle birlikte, bu mikroorganizmaların yavaş üremesi, kültür yöntemlerinin zor ve zahmetli olması ve uzun zaman alması gibi dezavantajları nedeniyle hızlı tanı yöntemlerine gereksinim artmaktadır. Günümüzde; tüberküloz basilinin hızlı üremesini ve erken dönemde tespit edilmesini sağlamayı amaçlayan, BACTEC gibi hızlı sonuç veren kültür sistemleri, mikobakteri laboratuvarlarının rutinleri arasına girerek bu süreyi kısaltmıştır. Ancak MGIT pozitif kültürlerden mikobakterilerin kesin tanısı için yapılan biyokimyasal testler zor ve zaman alıcı olmakta, birden fazla testin bir arada yapılması gerekmekte ve bu testlerle yapılan tanımlama çoğu zaman yetersiz kalmaktadır4,28.
FISH yöntemi, hedef genin izolasyon ve amplifikasyonuna gereksinim duymadan, karışık türler içeren ortamlarda bakterilerin tür düzeyinde tanımlanmasına olanak sağlayan hızlı, pratik ve ucuz bir yöntem olması nedeniyle diğer moleküler yöntemlere göre bazı avantajlar sunmaktadır. PNA-FISH ve kültür yöntemleriyle kıyaslandığında; Oligo-FISH yönteminin en önemli avantajı ucuz olmasıdır. Bununla birlikte bu yöntemin; a) özellikle mikobakteri türlerine özgül mevcut prob sayısının az olması, b) bazı prob ve bakteri türleri için optimizasyon gerektirmesi, c) tüm solüsyonların laboratuvar tarafından hazırlanması gerekliliği gibi bazı kısıtlılıkları bulunmaktadır10,11,12,13.
PNA-FISH, rRNA'yı hedef alan floresan işaretli PNA problar kullanılarak uygulanan ve son yıllarda kullanıma giren yeni bir FISH yöntemdir. PNA problar DNA'ya bağlanma kabiliyetinde olan psödopeptidlerdir; kısmen hidrofobik özellik gösterirler ve hem DNA hem de RNA ile hibridize olurlar. PNA probların avantajları arasında; a) yüksek afinite ve özgüllüğe sahip olması, b) hidrofobik hücre duvarından kolaylıkla difüze olması, c) uzun fiksasyon işlemlerine ihtiyaç olmaması ve hücre morfolojisini bozmaması, d) rutin klinik mikobakteriyoloji laboratuvarlarının gereksinimini karşılayabilecek düzeyde Mycobacterium spp. (MTC, NTM, M.immunogenum, M.leprae, M.avium, M.kansasii gibi) için özgül probların bulunması, e) kullanılan solüsyonların ve probların hazır olarak sunulması, sayılabilir14,15,16,17,18. Bu yöntemin önemli dezavantajı ise, ticari bir kitin henüz olmaması ve oligo-FISH'e göre daha pahalı olmasıdır. Son yıllarda diğer moleküler yöntemlere paralel olarak geliştirilen yeni FISH teknikleri tıbbi genetik ve mikrobiyal ekoloji gibi diğer alanlarda yaygın kullanım alanı bulmakla birlikte10, bu teknikler ülkemizde klinik mikrobiyoloji laboratuvarlarında yer alamamıştır. Bu çalışmada, mikobakteriyoloji laboratuvarında MGIT pozitif kültürlerden mikobakterilerin tanımlanması için iki farklı FISH yönteminin rutin laboratuvar kullanımındaki tanısal performansı değerlendirilmiştir.
Çalışma sonuçlarımız, MGIT pozitif kültürlerden mikobakterilerin tanımlanmasında her iki FISH yönteminin de konvansiyonel yöntemlerle mükemmel uyum gösterdiğini; her iki yöntemin de duyarlılık, özgüllük, pozitif ve negatif prediktif değerlerinin yüksek olduğunu göstermiştir. Yapılan diğer çalışmalarda da, bu yöntemlerin duyarlılık ve özgüllüklerinin yüksek olduğu bildirilmiştir15,29. FISH yönteminin en önemli avantajı diğer moleküler yöntemlere göre ucuz olmasıdır. Maliyet yönünden karşılaştırıldığında; Oligo-FISH'in PNA-FISH'e göre daha ucuz olduğu saptanmıştır. Bununla birlikte Oligo-FISH yönteminde kullanılacak probların sentezi, hibridizasyon için prob ve solüsyonların hazırlanması ve yöntemin optimizasyonu, laboratuvarların kendileri tarafından yapılmasını gerektirmektedir. PNA-FISH yönteminin, prob ve solüsyonların hazır olarak sunulması ve ayrıca MTC ve NTM gibi daha özgül tanımlamaya olanak sağlaması yönünden avantajları bulunmaktadır.
Çalışmamızda her iki yöntemde de bazı referans mikobakteri suşlarının CY3/FITC işaretli problarla aynı preparatta farklı floresan yoğunluğuna sahip oldukları görülmüştür. M.tuberculosis, M.abcessus, M.vaccae ve M.aurum daha güçlü floresan sinyal verirken; M.gordonae, M.senegalense ve M.scrofulaceum aynı preparatta kord oluşumu içinde zayıf, dışında ise daha güçlü floresan sinyal vermiştir. Mikobakterilerde prob sinyal yoğunluğundaki farklılıklar, bakterilerin farklı üreme koşulları ve farklı aktivitede olmaları ile ilgili olabileceği gibi, bakterilerin ribozom içeriği, hücre duvar yapısındaki mikolik asit kompozisyonu ile ilgili türlere ait değişimler veya kord oluşumu gibi pek çok faktöre de bağlı olabilir2,9,13,30,31. Yapılan bazı çalışmalarla, mikobakterilerin mikolik asit içeren hidrofobik yapısından oligonükleotid probların geçişini kolaylaştırmak için farklı kimyasallar (Etanol, PFA, HCl) ile fiksasyon ve çeşitli enzim ve kimyasallar (mutanolizin, lizozim, lipaz, proteinaz K, HCl) kullanılarak fiksasyon sonrası işlemleri ile yöntem modifiye edilmeye çalışılmıştır13,20,31,32.. Çalışmamızda, MGIT pozitif kültürlerden elde edilen fikse edilmiş örneklerde Oligo-FISH yöntemi daha önce Amann24 tarafından tarif edilen ve De Los Reyes ve arkadaşlarının20 minör modifikasyonlar eklediği yöntem esas alınmış ve küçük değişiklikler yapılmıştır. Çalışma sonuçları, MGIT pozitif kültürlerden mikobakteri türlerinin tanımlanmasında yöntemler arasında önemli bir farklılık olmadığını göstermiştir. Referans mikobakteri suşlarındaki türlere ait bireysel farklılıklarda, yöntem daha kapsamlı olarak ele alınıp diğer modifikasyonlar denenebilir.
FISH yönteminde yapılan modifikasyonlarla yöntemin duyarlılık ve özgüllüğü artmaktadır. Rodriguez-Nuñez ve arkadaşlarının33 lenf nodu örneklerinde modifiye FISH yöntemi ile yaptıkları çalışmada, yöntemin duyarlılık ve özgüllüğünün arttığı bildirilmiştir. Bu araştırmacılar, modifiye FISH yönteminin duyarlılık ve özgüllüğünü sırasıyla %71.4 ve %84.6; PCR yönteminin ise sırasıyla %62.5 ve %77.8 olarak bulmuşlar; direkt mikroskobik yöntemlerin duyarlılık ve özgüllüğünü moleküler yöntemlerden daha düşük olarak saptamışlardır33. Çalışmamızda MGIT pozitif kültürlerden mikobakterilerin tanımlanmasında, Oligo-FISH ile pozitif bulunan beş örnek PNA-FISH ile negatif bulunmuş, ancak daha sonra çalışma protokolündeki minör modifikasyonla bu örnekler de PNA-FISH ile pozitif saptanarak MTC veya NTM olarak tiplendirilmişlerdir. Bu durum az sayıda da olsa negatif örneklerin genel bir probla doğrulanması veya fiksasyon aşamasında modifikasyon yapılması gerektiğini göstermiştir. Bu çalışmada PNA-FISH'in diğer dezavantajı ise negatif kontrol probun olmamasıdır; ancak bu sorun da yeni prob geliştirilmesiyle veya Oligo-FISH'in birlikte kullanımı ile çözülebilir. PNA-FISH'in Oligo-FISH'e göre bazı üstünlükleri (kolay, tür düzeyinde identifkasyon vb.) olmasına rağmen, uygulamada Oligo-FISH ile birlikte kullanılması hem maliyeti azaltacak hem de pozitif örneklerin gözden kaçırılmasını önleyecektir. Sonuçlarımız ayrıca çalışmada kullanılan bazı probların SILVA veri tabanındaki mikobakterilerin tamamını kapsamadığını göstermektedir. Ancak MGIT pozitif kültürlerle yapılan bu çalışmada, klinik örneklerde saptanan mikobakterilerin bu problarla saptanmasında sorun olmadığı görülmüştür. Bununla birlikte probların zaman içinde güncellenmesi veya tür düzeyinde daha özgül probların geliştirilmesi gerekli olabilir.
Sonuç olarak, duyarlılık ve özgüllüğü yüksek olan PNA- ve Oligo-FISH yöntemlerinin, standardizasyonun sağlanması koşuluyla, mikobakteriyoloji laboratuvarlarında mikobakterilerin hızlı ve doğru tanımlanmasında uygulanabilecek yöntemler olduğu kanısına varılmıştır.
KAYNAKLAR
- World Health Organization. Global Tuberculosis Report 2013. Available at: http://apps.who.int/iris/bitstream/10665/91355/1/9789241564656_eng.pdf
- Tille PM (ed). Mycobacteria, pp: 484-510. In: Bailey & Scott's Diagnostic Microbiology. 2013, 13th ed. Elsevier, Mosby, St.Louis.
- Palomino JC. Nonconventional and new methods in the diagnosis of tuberculosis: feasibility and applicability in the field. Eur Respir J 2005; 26(2): 339-50.
- Çetin ES, Aynali A, Öztürk Tuba, Özseven AG, Kaya S. Mikobakterilerin klinik ömeklerden izolasyonunda Löwenstein-Jensen besiyeri kültürü ve Bactec Mycobacterium Growth Indicator Tube 960 sisteminin değerlendirilmesi. S.D.Ü. Tıp Fak Derg 2012; 19(1): 12-6.
- Neonakis IK, Gitti Z, Krambovitis E, Spandidos DA. Molecular diagnostic tools in mycobacteriology. J Microbiol Methods 2008; 75(1): 1-11.
- Soini H, Musser JM. Molecular diagnosis of mycobacteria. Clin Chem 2001; 47(5): 809-14.
- Wolff A, Perch-Nielsen IR, Poulsen CR, El-Ali J, Bang DD. Removal of PCR inhibitors using dielectrophoresis for sample preparation in a microfabricated system. 7th International Conference on Miniaturized Chemical and Biological Analysis Systems. October 5-9, 2003, Squaw Valley, California. Conference Book, p: 1137-40.
- Fredricks DN, Relman DA. Improved amplification of microbial DNA from blood cultures by removal of the PCR inhibitor sodium polyanetholesulfonate. J Clin Microbiol 1998; 36(10): 2810-6.
- Amann R, Fuchs BM. Single-cell identification in microbial communities by improved fluorescence in situ hybridization techniques. Nat Rev Microbiol 2008; 6(5): 340-8.
- Volpi EV, Bridger JM. FISH glossary: an overview of the fluorescence in situ hybridization technique. Biotechniques 2008; 45(4): 385-409.
- Stender H, Fiandaca M, Hyldig-Nielsen JJ, Coull J. PNA for rapid microbiology. J Microbiol Methods 2002; 48(1): 1-17.
- St. Amand AL, Frank DN, De Groote MA, Basaraba RJ, Orme IM, Pace NR. Use of specific rRNA oligonucleotide probes for microscopic detection of Mycobacterium tuberculosis in culture and tissue specimens. J Clin Microbiol 2005; 43(10): 5369-71.
- Davenport RJ, Curtis TP, Goodfellow M, Stainsby FM, Bingley M. Quantitative use of fluorescent in situ hybridization to examine relationships between mycolic acid-containing Actinomycetes and foaming in activated sludge plants. Appl Environ Microbiol 2000; 66(3): 1158-66.
- Drobniewski FA, More PG, Haris GS. Differentiation of Mycobacterium tuberculosis complex and nontuberculous mycobacterial liquid cultures by using peptide nucleic acid-fluorescence in situ hybridization probes. J Clin Microbiol 2000; 38(1): 444-7.
- Hongmanee P, Stender H, Rasmussen OF. Evaluation of a fluorescence in situ hybridization assay for differentiation between tuberculous and nontuberculous Mycobacterium species in smears of Lowenstein-Jensen and Mycobacteria Growth Indicator Tube cultures using peptide nucleic acid probes. J Clin Microbiol 2001; 39(3): 1032-5.
- Lefmann M, Schweickert B, Buchholz P, et al. Evaluation of peptide nucleic acid-fluorescence in situ hybridization for identification of clinically relevant mycobacteria in clinical specimens and tissue sections. J Clin Microbiol 2006; 44(10): 3760-7.
- Selvaraju SB, Kapoor R, Yadav JS. Peptide nucleic acid-fluorescence in situ hybridization (PNA-FISH) assay for specific detection of Mycobacterium immunogenum and DNA-FISH assay for analysis of pseudomonads in metalworking fluids and sputum. Mol Cell Probes 2008; 22(5-6): 273-80.
- Moter A, Göbel UB. Fluorescence in situ hybridization (FISH) for direct visualization of microorganisms. J Microbiol Methods 2000; 41(2): 85-112.
- Stender H, Mollerup TA, Lund K, Petersen KH, Hongmanee P, Godtfredsen SE. Direct detection and identification of Mycobacterium tuberculosis in smear-positive sputum samples by fluorescence in situ hybridization (FISH) using peptide nucleic acid (PNA) probes. Int J Tuberc Lung Dis 1999; 3(9): 830-7.
- De Los Reyes FL, Ritter W, Raskin L. Group-specific small-subunit rRNA hybridization probes to characterize filamentous foaming in activated sludge systems. Appl Environ Microbiol 1997; 63(3): 1107-17.
- Schuppler M, Wagner M, Schön G, Göbel U. In situ identification of nocardioform actinomycetes in activated sludge using fluorescent rRNA-targeted oligonucleotide probes. Microbiology 1998; 144(1): 249-59.
- Amann RI, Binder BJ, Olson RJ, Chisholm SW, Devereux R, Stahl DA. Combination of 16S rRNA-targeted oligonucleotide probes with flow cytometry for analyzing mixed microbial populations. Appl Environ Microbiol 1990; 56(6): 1919-25.
- Wallner G, Amann R, Beisker W. Optimizing fluorescent in situ hybridization with rRNA-targeted oligonucleotide probes for flow cytometric identification of microorganisms. Cytometry 1993; 14(2): 136-43.
- Amann RI. In situ identification of micro-organisms by whole cell hybridization with rRNA-targeted nucleic acid probes, pp: 1-15. In: Akkermans ADL, van Elsas JD, De Bruijn FJ (eds), Molecular Microbial Ecology Manual 3.3.6. 1995. Kluwer Academic Publishers, Dordrecht, The Netherlands.
- Lawrence JB, Singer RH. Quantitative analysis of in situ hybridization methods for the detection of actin gene expression. Nucleic Acids Res 1985; 13(5): 1777-99.
- Lee LV. Procedures for Identification From Culture: Mycobacteriology and Antimycobacterial Susceptibility Testing, pp: 7.6.1.1-12. In: Isenberg HD (ed), Clinical Microbiology Procedures Handbook. 2004. ASM Press, Washington, DC.
- Weitzman I. Acid-Fast Stains: Mycobacteriology and Antimycobacterial Susceptibility Testing, pp: 7.2.1.1-4. In: Isenberg HD (ed), Clinical Microbiology Procedures Handbook. 2004. ASM Press, Washington, DC.
- Özakın C, Gedikoğlu S. Tüberküloz Tanısında Tüberküloz Laboratuvarının Rolü: Tanı ve İlaç Duyarlılık Testlerinde Rutin Laboratuvar Yöntemlerinin Değeri. 21. Yüzyılda Tüberküloz Sempozyumu ve II. Tüberküloz Laboratuvar Tanı Yöntemleri Kursu. 11-12 Haziran 2003, Samsun. Kurs Kitabı, s: 397-401.
- Stender H, Lund K, Petersen KH, et al. Fluorescence in situ hybridization assay using peptide nucleic acid probes for differentiation between tuberculous and nontuberculous mycobacterium species in smears of mycobacterium cultures. J Clin Microbiol 1999; 37(9): 2760-5.
- Tomiyasu I. Mycolic acid composition and thermally adaptive changes in Nocardia asteroides. J Bacteriol 1982; 151(2): 828-37.
- Carr EL, Eales K, Soddell J, Seviour RJ. Improved permeabilization protocols for fluorescence in situ hybridization (FISH) of mycolic-acid-containing bacteria found in foams. J Microbiol Methods 2005; 61(1): 47-54.
- Macnaughton SJ, O'Donnell AG, Embley TM. Permeabilization of mycolic-acid-containing actinomycetes for in situ hybridization with fluorescently labelled oligonucleotide probes. Microbiology 1994; 140(Pt 10): 2859-65.
- Rodriguez-Nuñez J, Avelar FJ, Marquez F, Rivas-Santiago B, Quiñones C, Guerrero-Barrera AL. Mycobacterium tuberculosis complex detected by modified fluorescent in situ hybridization in lymph nodes of clinical samples. J Infect Dev Ctries 2012; 6(1): 58-66.
İletişim (Correspondence):
Doç. Dr. Gülay Börekçi,
Mersin Üniversitesi Sağlık Yüksekokulu,
Çiftlikköy Kampüsü, Mersin, Türkiye.
Tel (Phone): +90 324 361 0001/4223,
E-posta (E-mail): gulay_borekci@yahoo.com