Klinik Örneklerden İzole Edilen Trimetoprim-Sülfametoksazole Dirençli
Stenotrophomonas maltophilia Suşlarında İntegron, sul1-2 ve dfr Genlerinin Araştırılması*
Investigation of Integrons, sul1-2 and dfr Genes in Trimethoprim-Sulfametoxazole-Resistant
Stenotrophomonas maltophilia Strains Isolated from Clinical Samples
Esra ÖZKAYA1, Faruk AYDIN2, Gülçin BAYRAMOĞLU2, Celal Kurtuluş BURUK2, Cemal SANDALLI3
1 Kahramanmaraş Necip Fazıl Şehir Hastanesi, Mikrobiyoloji Laboratuvarı, Kahramanmaraş.
1 Kahramanmaraş Necip Fazıl City Hospital, Microbiology Laboratory, Kahramanmaraş, Turkey.
2 Karadeniz Teknik Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Trabzon.
2 Karadeniz Technical University Faculty of Medicine, Department of Medical Microbiology, Trabzon, Turkey
3 Recep Tayyip Erdoğan Üniversitesi, Fen-Edebiyat Fakültesi, Biyoloji Bölümü, Rize.
3 Recep Tayyip Erdoğan University Faculty of Arts and Sciences, Department of Biology, Rize, Turkey.
* Bu çalışma, XXXIV. Türk Mikrobiyoloji Kongresi (07-11 Kasım 2010, Girne, KKTC)'nde poster olarak sunulmuştur.
ÖZET
Nonfermentatif gram-negatif basil olan Stenotrophomonas maltophilia, hastane enfeksiyonları ve fırsatçı enfeksiyonlar açısından giderek artan bir öneme sahiptir. Çeşitli mekanizmaları kullanarak aminoglikozidler, beta-laktamlar, tetrasiklinler gibi birçok geniş spektrumlu antibiyotiğe direnç gösterir. Bu durum klinisyenlerin ampirik tedavi seçeneklerini oldukça kısıtlayabilmektedir. Trimetoprim-sülfametoksazol (SXT), hem yüksek etki gücü hem de geniş bir hasta yelpazesinde kullanılabildiği için S.maltophilia enfeksiyonlarının tedavisinde ilk tercih olarak önerilen antibiyotiktir. Ancak son yıllarda farklı coğrafi bölgelerde yapılan çalışmalarda bu antibiyotiğe karşı da direnç görüldüğü bildirilmeye başlanmıştır. Bu çalışmada SXT'ye dirençli S.maltophilia izolatlarında, bu dirence neden olduğu bilinen sul1, sul2, dfrA9, dfrA10, dfrA20 genlerinin ve sınıf I, II integron gen kasetlerinin araştırılması amaçlanmıştır. Çalışmaya, Karadeniz Teknik Üniversitesi Tıp Fakültesi, Farabi Hastanesi Tıbbi Mikrobiyoloji Anabilim Dalı Laboratuvarında, Ocak 2006-Ekim 2011 tarihleri arasında 339 hastaya ait çeşitli klinik örneklerden izole edilen 618 S.maltophilia suşu dahil edilmiştir. İzolatlar, konvansiyonel yöntemlere ilaveten Phoenix otomatize sistemi (Becton Dickinson, ABD) ile de tanımlanmış; otomatize sistem ve agar dilüsyon yöntemi ile 32 hasta izolatında (32/339, %9.4) SXT direnci saptanmıştır. Bu suşların 29 (%90.6)'u hastane, 3 (%9.4)'ü toplum kaynaklı enfeksiyonlardan izole edilmiştir. SXT-dirençli 32 farklı S.maltophilia izolatında dirence neden olduğu bilinen sul1, sul2, dfr genleri ve sınıf I ve II integron gen kasetleri özgül primerler kullanılarak polimeraz zincir reaksiyonu yöntemiyle araştırılmış, ardından amplifiye edilen ürünlerin nükleotid dizi analizleri yapılmıştır. Bu işlemlerin sonucunda bir izolatta sınıf I integron gen kaseti ve sul1 geninin varlığı tespit edilmiştir. Gen kasetinin nükleotid dizi analizi incelendiğinde, sırasıyla beta-laktamaz enzimlerinden biri olan oksasilinaz (oxa-2), aminoglikozid direncine neden olan aminoglikozid 6'-N-asetiltransferaz [aac(6')-IIc] ve kuaterner amonyum bileşikleri direnç genlerini (qacF) taşıdığı saptanmıştır. Sonuç olarak, S.maltophilia'da integron sınıf I gen kasetinin sahip olduğu oxa2, aac(6')-IIc ve qacF gen dizilimi, bizim bildiğimiz kadarıyla ilk kez tanımlanmıştır. İntegron sınıf I gen kaseti ve sul1 geninin birlikteliğinin, S.maltophilia'da çoğul antibiyotik direnci gelişmesine aracılık edebileceği ve direncin yayılmasında kaynak oluşturabileceği göz önünde bulundurulmalıdır.
Anahtar sözcükler: Stenotrophomonas maltophilia; trimetoprim-sülfametoksazol; direnç; integron; gen kaseti.
ABSTRACT
Stenotrophomonas maltophilia, which is a non-fermentative gram-negative bacillus, has an increasing importance in nosocomial and opportunistic infections. Since it exhibits resistance to numerous broad-spectrum antibiotics such as aminoglycosides, beta-lactams and tetracyclines, it may considerably limit empirical treatment options. Trimethoprim-sulfamethoxazole (SXT) is recommended as the first-line therapy in the treatment of S.maltophilia infections thanks to its high potency and usefulness in a range of patients. In recent years, however, studies in different geographical regions have started to report resistance to SXT. In this study, we aimed to investigate the genes sul1, sul2, dfrA9, dfrA10, dfrA20 and class I, class II integron gene cassettes which are known to play role in SXT resistance among SXT-resistant S.maltophilia strains. A total of 618 S.maltophilia strains isolated from various clinical samples of 339 patients between January 2006 and October 2011 at the laboratory of Medical Microbiology Department, Faculty of Medicine, Karadeniz Technical University, Trabzon, Turkey, were included in the study. The isolates were identified by both conventional methods and the Phoenix automated identification system (Becton Dickinson, USA). SXT resistance was determined in the isolates of 32 patients (32/339, 9.4%) by both the automated system and agar dilution method of them 29 (90.6%) were hospital-acquired, and 3 (9.4%) were community-acquired. The genes which are known as SXT resistance determining genes including sul1, sul2, dfr genes, and class I and class II integron gene cassettes were analyzed by using specific primers with polymerase chain reaction in the 32 SXT-resistant isolates. Subsequently, nucleotide sequence analysis of the amplified materials was performed. As a result of this assay, the presence of class I integron gene cassette and sul1 gene were detected in one isolate. Nucleotide sequence analysis of the gene cassette revealed oxacilinase (oxa2) type of beta-lactamase, an aminoglycoside 6'-N-acetyltransferase [aac(6')-IIc], leading to resistance of aminoglycosides, and a quaternary ammonium compounds resistance gene (qacF), respectively. In conclusion, to best of our knowledge the sequences of class I integron gene cassette including oxa2, aac(6')-IIc, qacF genes were identified in S.maltophilia for the first time. It should be kept in mind that the co-presence of a class I integron gene cassette and the sul1 gene in S.maltophilia may lead to the development of multi-drug resistance and may act as a potential source for the dissemination of resistance.
Key words: Stenotrophomonas maltophilia; trimethoprim-sulfamethoxazole; resistance; integron; gene cassette.
Geliş Tarihi (Received): 22.11.2013 • Kabul Ediliş Tarihi (Accepted): 02.03.2014
GİRİŞ
İlk kez 1943 yılında plevral sıvıdan izole edilen Stenotrophomonas maltophilia'nın hastane enfeksiyonları ve fırsatçı enfeksiyonlar açısından önemi giderek artmaktadır1. Hastanede yatış süresinin uzaması, yoğun bakım ünitelerinde yatış, kronik solunum yolu hastalıkları (kistik fibrozis gibi), geniş spektrumlu antibiyotik kullanımı, malignansiler, immün süpresyon, mukokütanöz bariyerin bozulması (mekanik ventilasyon, kateter girişimleri, trakeotomi, periton diyalizi vb.), prematürite gibi risk faktörleri taşıyanlarda S.maltophilia enfeksiyonlarının görülme sıklığı yüksektir2.
S.maltophilia, dış membran geçirgenliğinde azalma, efluks pompa genlerinin ekspresyonunu artırma, çeşitli enzimlerin üretimi (aminoglikozid modifiye eden enzim, beta-laktamaz vb.) gibi mekanizmaların varlığı nedeniyle aminoglikozidler, beta-laktamlar, tetrasiklinler gibi pek çok geniş spektrumlu antibiyotiğe direnç gösterir. Bu durum klinisyenlerin ampirik tedavi seçeneklerini oldukça kısıtlamaktadır3,4. Son yıllarda farklı coğrafi bölgelerde yapılan çalışmalarda, hem yüksek etki gücü hem de kullanılabilen hasta profilinin genişliği nedeniyle tedavide ilk tercih olarak önerilen trimetoprim-sülfametoksazole (SXT) karşı direnç geliştiği bildirilmeye başlanmıştır5,6,7. S.maltophilia'nın horizontal geçişle gram-pozitif bakterilerden bile antibiyotik direnç genleri alabileceği gösterilmiştir. İntegronlar bu aktarımda önemli rol oynamaktadır. Özellikle sul1 veya sul2 sülfonamid direnç genlerini taşıyan suşlar son zamanlarda sıkça rapor edilmektedir. Benzer şekilde bir aktarımla, dihidrofolat enzim inhibisyonu yapan dfrA genlerini elde etmesiyle trimetoprime karşı da direnç kazanabilmektedir3,6,8,9. Bu çalışmada, Karadeniz Teknik Üniversitesi Tıp Fakültesi Farabi Hastanesi Tıbbi Mikrobiyoloji Anabilim Dalı Laboratuvarında, Ocak 2006-Ekim 2011 tarihleri arasında çeşitli klinik örneklerden izole edilen SXT'ye dirençli S.maltophilia izolatlarında, bu dirence neden olduğu bilinen sul1, sul2, dfrA9, dfrA10, dfrA20 genlerinin ve sınıf I, II integron gen kasetlerinin varlıklarının araştırılması amaçlanmıştır.
GEREÇ ve YÖNTEM
İzolatlar
Çalışmaya, Ocak 2006-Ekim 2011 tarihleri arasında laboratuvarımızda farklı klinik örneklerden izole edilen S.maltophilia izolatları dahil edildi. Bu süreç içinde 339 hastaya ait 618 örnekten S.maltophilia tanımlandı ve bunların içinden SXT'ye dirençli kökenler seçildi. Bu seçimde her hastanın ilk izolatı olması baz alındı. İnceleme sonucunda toplam 32 adet SXT'ye dirençli S.maltophilia izolatında antibiyotik direnç genleri araştırıldı. İşlemler uygulanıncaya kadar izolatlar %15 gliserol (Merck, Almanya) içeren triptik soy buyyon besiyerinde (Fluka, 22092, ABD) -80ºC'de saklandı10.
Klinik örnekler, %5 koyun kanlı agar (Salubris, Türkiye), EMB (Eosin-Methylene-Blue) agar (Oxoid, İngiltere) ve çikolatamsı agar (Salubris, Türkiye) besiyerlerine ekilerek 24-48 saat 37ºC'de aerobik ortamda inkübe edildi ve üreyen koloniler değerlendirilmeye alındı. Boyalı mikroskobik incelemede gram-negatif basil görünümünde olan katalaz pozitif, oksidaz negatif, nonfermentatif bakteriler klasik yöntemlere ilaveten Phoenix otomatize bakteri tanımlama ve antibiyotik duyarlılık sistemi (Becton Dickinson Diagnostic Instrument Systems, Sparks, ABD) ile üretici firma önerileri doğrultusunda tür düzeyinde tanımlandı.
Antimikrobiyal Duyarlılık Testleri ve Minimum İnhibitör Konsantrasyonu (MİK) Tayini
S.maltophilia olarak tanımlanan izolatların antibiyotik duyarlılıkları, NMLC/ID35 kullanılan BD Phoenix sisteminin yanı sıra Kirby-Bauer metoduna göre Mueller-Hinton agar (MHA) kullanılarak standart disk difüzyon yöntemiyle de araştırıldı11. SXT duyarlılığı agar dilüsyon yöntemi ve Phoenix otomatize sistemiyle; seftazidim ve levofloksasin duyarlılığı Phoenix otomatize sistemiyle; minosiklin duyarlılığı ise disk difüzyon yöntemi ile çalışıldı. Sonuçlar, CLSI (Clinical and Laboratory Standards Institute) kriterlerine göre duyarlı ve dirençli olarak kategorize edildi12,13.
Otomatize duyarlılık sistemi sonucuna göre SXT'ye dirençli bulunan izolatların MİK değerleri agar dilüsyon yöntemiyle belirlendi. Literatürdeki bildirimler ve kalite kontrol suşlarının değer aralıklarını kapsayacak şekilde SXT konsantrasyonları 0.06/1.19- 64/1216 µg/ml arasında olan 11 farklı dilüsyon içeren MHA besiyeri hazırlandı. Kalite kontrol suşu olarak Escherichia coli ATCC 24922 ve Pseudomonas aeruginosa ATCC 27853 suşları kullanıldı. Her deneyde üremenin kontrolü için herhangi bir antimikrobiyal ajan içermeyen MHA besiyerine (antimikrobiyal içeren besiyerlerine ekim öncesi ve sonrasında olacak şekilde) aynı şekilde inokülasyon yapıldı. Tüm besiyerleri 37ºC'de 24-48 saat inkübe edildikten sonra gözle görünür koloni oluşumu not edilerek her izolat için MİK değerleri saptandı14.
DNA İzolasyonu ve Polimeraz Zincir Reaksiyonu (PCR)
Stok ortamından alınan suşlar önce %5 koyun kanlı agara ekildi. Üreyen bakterilerin Luria Bertani (LB) sıvı besiyerine tek koloni pasajları yapıldıktan sonra çalkalamalı su banyosunda bir gece inkübe edildi. Bu süspansiyondan 1.5 ml alınarak 5000 rpm'de 5 dakika santrifüj edilip süpernatanı atıldı. Çökelti bir kez 1 ml Tris-EDTA tamponu, ardından da bir kez 1 ml steril distile su ile yıkandı. Kalan çökeltiye 500 µl steril distile su eklenerek 15 dakika kaynayan suda bekletildi. Kaynatma işleminden sonra 13.000 rpm'de 5 dakika santrifüj edildi. Üstte kalan süpernatan kısmından 400 µl alınıp ayrı bir mikrosantrifüj tüpüne aktarıldı. Böylece PCR işleminde kullanılacak kalıp DNA'lar elde edildi15. PCR yönteminde kullanılan primer dizileri (IDT, ABD ve Biomers, Almanya) Tablo I'de gösterildi.
Elektroforez işlemi için %2 ve %1'lik hazırlanan agaroz jele son konsantrasyonu 0.5 μg/ml olacak şekilde etidyum bromür eklendi. Ürün büyüklüklerine göre 1 kb DNA belirteci (MBI Fermentas, ABD) veya 100 baz çifti (bç) DNA belirteci (New England BioLabs, İngiltere) ilk ve son kuyucuklara 5 µl pipetlendi. Elektroforez işleminde oluşan bantlar UV transilüminatörde gözlendi ve VersaDoc Imaging System (Bio-Rad, ABD) ile görüntülendi.
Nükleotid Dizi Analizi
Çalışma kapsamında kullanılan E.coli JM101 kökenine ait kompetan hücreler kalsiyum klorür metodu ile hazırlandı. Baz sırasının belirlenmesi için PCR ürünlerinin pGEM-T easy vektörüne (Promega, ABD) ligasyonu sağlandı ve bu amaçla üretici firmaya ait protokol izlendi. Ligasyon ürünleri daha önceden hazırlanan E.coli JM101 kompetan hücrelerine transforme edildi19. PCR ürününü taşıyan plazmidi içeren hücreler 1 mM IPTG ve X-Gal içeren ampisilinli (50 μg/ml) LB agar petrilerine yayma ekim ile ekildi ve mavi-beyaz renk oluşumuna bakılarak hücreler ayrıldı. Yirmi koloni beyaz hücreden plazmid izole edildi (Wizard Plus SV Minipreps DNA Purification System, Promega, ABD) ve EcoRI restriksiyon endonükleaz enzimi ile kesilerek agaroz jelde yürütüldü. Kontrol olarak PCR ürünü kullanıldı ve PCR ürünü ile aynı parçayı veren plazmidler pozitif olarak kabul edilerek nükleotid dizi analizi için Macrogen Inc.'e (Amsterdam, Hollanda) gönderildi.
Ters primer kullanılarak elde edilen dizilere ait çakışma bölgeleri, BLAST programı (URL-1, 2005) kullanılarak belirlendi. Üst üste çakışan kısımlar alt alta getirilerek birleştirildi ve gene ait tam uzunlukta dizi elde edildi.
BULGULAR
Çalışmamızda, 339 hastanın farklı klinik örneklerden izole edilen 618 S.maltophilia izolatı incelenmiş; bunlardan SXT'ye dirençli bulunan 32 (%9.4)'si çalışmaya alınmıştır. İzolatların 29 (%90.6)'u hastane kaynaklı, 3 (%9.4)'ü toplum kaynaklıdır. İzolatların klinik birimlere ve örneklere göre dağılımı Tablo II'de, antibiyotik duyarlılık sonuçları ise Tablo III'te görülmektedir.
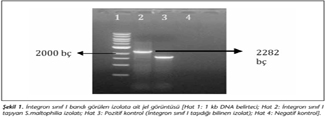
Direnç genlerinin araştırılması sonucunda, intl1 ve intl2 primerleri ile yapılan amplifikasyon sonucunda 32 hasta izolatının 2'sinde 500 bç büyüklüğünde bant saptanmış; 5'CS ve 3'CS primerleriyle yapılan integron sınıf I taramasında sadece bir izolatta 2282 bç büyüklüğünde bant görülmüş (Şekil 1), diğer izolatta bant görülmemiş; hep51 ve hep74 primerleriyle integron sınıf II taraması sonucunda ise hiçbir izolatta bant tespit edilmemiştir.
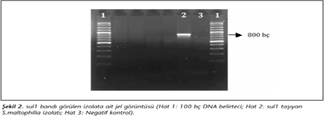
sul1 primerleri ve PCR koşulları ile yapılan sülfonamid direnci taraması sonucunda, sadece integron sınıf I gen kaseti bulunan izolatta 840 bç büyüklüğünde bir bant görülmüştür (Şekil 2). Elimizde bu gen dizisini içeren pozitif kontrol suşu bulunmadığı için görüntülenen bandın sul1 olduğu nükleotid dizi analizi ile doğrulanmıştır. sul2 primerleri ile yapılan taramada pozitif sonuç bulunamamıştır. SXT direncinin diğer bir nedeni olan dfr geni ise Tablo I'de verilen primerlerle ve kaynakta belirtilen PCR koşullarına uygun olarak taranmış ancak pozitif sonuç alınamamıştır.
TARTIŞMA
S.maltophilia; doğada kaynak suları, musluk suları, toprak, bitkiler gibi pek çok ortamda bulunabilen; hastane ortamında ise mortalite ve morbiditeyi artıran enfeksiyonlara neden olan önemli patojenlerden biridir1. Toplum kaynaklı enfeksiyonlara da yol açmasına karşın, asıl olarak hastane kaynaklı enfeksiyonlara neden olmaktadır20,21,22. Hastane ortamında musluk başları, banyolar gibi genel kullanım alanları dışında ultrasonografik görüntüleme cihazlarının başlıkları ve bu işlemde kullanılan jeller, diyalizatörler aparatları gibi malzemelerde üreyerek kontaminasyonlara yol açabilmektedir. Bizim çalışmamızdaki SXT'ye dirençli S.maltophilia suşlarının da çoğu (%90.6) hastane kaynaklıdır. S.maltophilia, hastanelerde, özellikle yoğun bakım üniteleri (YBÜ)'nde, uzun süreli yatan olgularda enfeksiyon oluşturma eğilimindedir2. Çalışmamızda, 2006-2011 yılları arasında izole edilen 618 S.maltophilia suşunun retrospektif değerlendirmesinde, izolatların en sık YBÜ'de (%39.8) saptandığı görülmüş; ileri incelemeye alınan SXT'ye dirençli S.maltophilia izolatlarının da %49.9'u yine YBÜ'de saptanmıştır.
Literatürdeki veriler daha çok erişkin popülasyona ait olsa da, S.maltophilia, yenidoğan ve pediyatrik yaş grubu için görülme sıklığı artan hastane enfeksiyonu etkenleri arasındadır. SENTRY Antimikrobiyal Sürveyans Programı tarafından 2004 yılında yapılan araştırmada, pediyatrik yaş grubundan etken patojen olarak izole edilen kökenler arasında S.maltophilia Latin Amerika'da 10., Kuzey Amerika'da 13. sırayı almış; Avrupa'da ise patojen olarak görülmemiştir23. Ülkemizde bu denli kapsamlı yapılmış bir araştırma bulunmamaktadır; ancak çalışmamızda hem SXT'ye dirençli hem de duyarlı S.maltophilia üretilen hasta grupları incelendiğinde, büyük kısmını yenidoğan YBÜ'deki bebeklerin oluşturduğu göze çarpmaktadır. Bizim çalışmamıza benzer şekilde, Mutlu ve arkadaşları24 2006-2008 yılları arasında hastanemiz yenidoğan YBÜ'de yaptıkları vaka-kontrol çalışmasında 46 klinik örnekte S.maltophilia izole etmişler ve bu yaş grubunda hayati enfeksiyonlara yol açabileceğini vurgulamışlardır.
S.maltophilia, solunum yolu enfeksiyonu, endokardit, üriner sistem enfeksiyonları, endoftalmit, yara ve yumuşak doku enfeksiyonları gibi pek çok klinik tabloya yol açabilmektedir22. Çalışma sürecinde izolasyon yapılan tüm örnekler retrospektif olarak incelendiğinde %58 ile ilk sırayı solunum yolu örneklerinin aldığı göze çarpmaktadır. Gülmez ve arkadaşları5 da, bizimle uyumlu olarak en sık solunum yolu örneklerinden S.maltpohilia izole etmişlerdir. Kendi hastanemizin verilerine bakıldığında ise Çaylan ve arkadaşları25 ilk sırada kan daha sonra trakeal aspirat kültürlerinde S.maltophilia ürettiklerini bildirmişlerdir. Sonuçlar karşılaştırıldığında, mikroorganizmanın üretildiği enfeksiyon bölgelerinde yıllar içinde büyük farklılıklar olmadığı gözlenmektedir. Bu durum, çalışmamızda incelenen SXT'ye dirençli S.maltophilia kökenleri için de değişmemiş ve en sık izolasyon, solunum yolu örneklerinden yapılmıştır.
S.maltophilia, karbapenemler gibi geniş spektrumlu pek çok antibiyotiğe doğal direnç göstermektedir1. Taşıdığı intrensek dirençlere ek olarak, her geçen gün dünyanın farklı coğrafi bölgelerinden kazanılmış direnç raporları bildirilmektedir6,9,23,26. Kazanılmış dirençle ilgili olarak çoğunlukla mobil gen bölgeleri suçlanmaktadır. Bu konuda son yıllarda kendilerine komşu genleri yanında taşıyabilen "insertion sequences common regions (ISCR)" elemanları gündeme gelmeye başlamıştır27. Bu durum klinisyenlerin elinde S.maltophilia tedavisinde kullanabilecekleri az sayıda ajan kalmasına neden olmaktadır. S.maltophilia enfeksiyonlarının tedavisinde ilk tercih edilmesi gereken ilacın SXT olduğu bildirilmekle birlikte, bu ajana karşı da direnç geliştiği yönünde raporlar giderek artmaktadır6,21,23. Çalışmamızda 339 hastaya ait S.maltophilia izolatı incelenmiş ve %9.4'ünün SXT'ye dirençli olduğu görülmüştür. Sader ve Jones'un7 SENTRY Antimicrobial Surveillance Programı (1997-2003) dahilinde yaptıkları çalışmada, tüm dünyada çeşitli örneklerden toplanan 3059 nonfermentatif gram-negatif basilin (Pseudomonas aeruginosa ve Acinetobacter spp. hariç) en büyük kısmını %59.2 (2076) ile S.maltophilia izolatları oluşturmuştur ve bu izolatların %4.7'sinde SXT direnci saptanmıştır. Ülkemizde bu konuda yapılan yayınlar incelendiğinde, Köseoğlu ve arkadaşları28 Hacettepe Üniversitesi Tıp Fakültesi Hastanesinde 1998-2001 yılları arasında çocuk hastalarda üreyen S.maltophilia izolatlarında SXT direncini %5 olarak saptamışlardır. Gülmez ve arkadaşları5 2005 yılındaki yayınlarında SXT direnç oranını %28.3 olarak bulmuşken, 2010 yılındaki yayınlarında29 %4 olarak tespit etmişlerdir. Tatman-Otkun ve arkadaşları30 2005 yılında yaptıkları çalışmada SXT direncini %1.9 olarak belirtmişlerdir. Hastanemiz verilerini incelersek Çaylan ve arkadaşları25 2004'te tüm yaş gruplarında ve tüm örneklerde S.maltophilia'da SXT direnç oranını %2.3, Mutlu ve arkadaşları24 yenidoğanlarda yaptıkları çalışmada %13 olarak tespit etmişlerdir. Çalışmamız sonucunda görüldüğü üzere tüm dünyada olduğu gibi bizim hastanemizde de S.maltophilia'da SXT direnci giderek önemli bir sorun halini almaktadır.
S.maltophilia suşlarında SXT direnci efluks pompalarının aşırı ekspresyonu veya biyofilm oluşumu gibi nedenlere bağlı olabileceği gibi, bizim çalışmamızda araştırdığımız mobil direnç genlerine bağlı enzimatik direnç de söz konusu olabilir. Lévesque ve arkadaşlarının17 kullandığı 3'CS ve 5'CS primerleriyle yaptığımız tarama sonucunda, 32 hasta izolatının sadece bir tanesinde integron sınıf I (int-I) geni tespit edilmiştir. Toleman ve arkadaşlarının6 kullandığı sul1-F ve sul1-R primerleriyle yapılan taramada ise, int-I tespit ettiğimiz izolatta sul1 geni de pozitif bulunmuştur. Bizim sonucumuza benzer şekilde Neela ve arkadaşları26 da 64 izolattan birinde int-I ve sul1 genlerini tespit etmişlerdir. Toleman ve arkadaşları6 tüm dünyadan toplanan 25 SXT'ye dirençli S.maltophilia izolatının 17'sinde int-I gen kasetini ve sul1 geninin birlikteliğini göstermişlerdir. Kore'de Song ve arkadaşları31 2007-2009 yılları arasında üç üniversite hastanesinden toplanan 120 S.maltophilia izolatının 19'unu SXT'ye dirençli bulmuş, bu izolatların 10'unda da int-I gen kaseti ve sul1 birlikteliğini saptamışlardır. Aynı durum Çin'de 2006-2009 yılları arasında 31 adet SXT'ye dirençli S.maltophilia izolatının 21'inde görülmüştür9.
Çalışmamızda bir izolatta PCR ile amplifiye ettiğimiz int-I gen kasetinin nükleotid dizi analizi incelendiğinde; beta-laktamaz geni (oxa2), aminoglikozid direnç geni [aac(6')-IIc] ve kuaterner amonyum direncine yol açan qacF geninin varlığı görülmüştür. Bu dizilimin hemen yanında ise sul1 geni bulunmaktadır. İntegronların aktarılabilir gen bölgeleri oldukları göz önünde tutulduğunda, tespit ettiğimiz gen kasetini taşıyan S.maltophilia izolatının direnç genleri açısından rezervuar rolü oynayabileceği düşünülmektedir. Benzer olarak, Malezya'da yapılan bir çalışmada, kuaterner amonyum direnç genleri bulunmuş ve bu genlerin SXT direnci oluşmasında etkili olduğu vurgulanmıştır26. Araştırıcılar, bu maddeyi içeren dezenfektanların önerilen dozların üzerinde kullanılması halinde, hastanelerde dirençli suşların seçiminin kolaylaşacağına ve direnç oranlarının artacağına dikkati çekmişlerdir26. SENTRY'nin 2007 yılında beş farklı kıtada yaptığı çalışmada, amplifiye edilen sınıf I integronların içerikleri; Almanya ve Brezilya izolatlarında sul ve qac, ABD ve Şili izolatlarında aacA4, Meksika'ya ait iki izolatta ise aacA7 ve aadA5 genleri olarak saptanmıştır6. Çin'de yapılan çalışmalarda ise, çoğunlukla aadA, aac, dfrA, cat genlerinin farklı birlikteliklerini içeren gen kasetleri tespit edilmiştir9,32. Kore'de aacA4, aadA1, aac6'-II ve qac genlerinden oluşan integron sınıf I gen kaseti bulunmuştur31. Tayvan'da yapılan bir çalışmada, bizim sonucumuza benzer direnç genleri (aacA4, aadB, aacC4, aacA6-Ib, smr, smr/aacA4, qac, cmlA, catB2 ve blaIMP-8/aac6-II/aadA5) saptansa da dizilimleri farklıdır33. Bütün bu incelemeler ile BLAST veri tabanı kullanılarak yapılan tarama sonucunda; çalışmamızda belirlenen integron sınıf I gen kasetinin sahip olduğu gen diziliminin ilk kez saptandığı görülmüştür.
Sonuç olarak bu çalışmada, ülkemizde S.maltophilia enfeksiyonlarının tedavisinde zorluk oluşturabilecek, mobil elemanlar üzerinde kodlanan ve enzimatik yolla SXT direncine yol açabilen sul1 geninin varlığı gösterilmiştir. Bu genin, bir integron ile birlikte mobilize olduğu dikkate alınırsa, direncin türler arasında da yayılabileceği düşünülmektedir. Hiçbir izolatta, araştırılan dfr genlerine rastlanmamıştır. sul1 saptanan izolatın dışındaki izolatlarda görülen fenotipik direncin nedenin efluks pompa sistemi, biyofilm oluşturma gibi farklı mekanizmalardan kaynaklanabileceği ya da henüz tanımlanmamış gen bölgelerinin varlığına bağlı olabileceği kanaatine varılmıştır.
KAYNAKLAR
- Denton M, Kerr KG. Microbiological and clinical aspects of infection associated with Stenotrophomonas maltophilia. Clin Microbiol Rev 1998; 11(1): 57-80.
- Brooke JS. Stenotrophomonas maltophilia: an emerging global opportunistic pathogen. Clin Microbiol Rev 2012; 25(1): 2-41.
- Nyc O, Matejková J. Stenotrophomonas maltophilia: Significant contemporary hospital pathogen-review. Folia Microbiol (Praha) 2010; 55(3): 286-94.
- Pages D, Rose J, Conrod S, et al. Heavy metal tolerance in Stenotrophomonas maltophilia. PLoS One 2008; 3(2): e1539.
- Gülmez D, Hasçelik G. Stenotrophomonas maltophilia: antimicrobial resistance and molecular typing of an emerging pathogen in a Turkish university hospital. Clin Microbiol Infect 2005; 11(11): 880-6.
- Toleman MA, Bennett PM, Bennett DM, Jones RN, Walsh TR. Global emergence of trimethoprim/sulfamethoxazole resistance in Stenotrophomonas maltophilia mediated by acquisition of sul genes. Emerg Infect Dis 2007; 13(4): 559-65.
- Sader HS, Jones RN. Antimicrobial susceptibility of uncommonly isolated non-enteric gram-negative bacilli. Int J Antimicrob Agents 2005; 25(2): 95-109.
- Sanchez MB, Hernandez A, Martinez JL. Stenotrophomonas maltophilia drug resistance. Future Microbiol 2009; 4(6): 655-60.
- Hu LF, Chang X, Ye Y, et al. Stenotrophomonas maltophilia resistance to trimethoprim/sulfamethoxazole mediated by acquisition of sul and dfrA genes in a plasmid-mediated class 1 integron. Int J Antimicrob Agents 2011; 37(3): 230-4.
- Robson RL, Essengue S, Reed NA, Horvat RT. Optochin resistance in Streptococcus pneumoniae induced by frozen storage in glycerol. Diagn Microbiol Infect Dis 2007; 58(2): 185-90.
- Clinical and Laboratory Standards Institute. Performance standards for antimicrobial disc susceptibility tests. Approved Standard, 9th ed. CLSI Document M2-A9, 2006. CLSI, Wayne, PA.
- O'Hara CM. Evaluation of the Phoenix 100 ID/AST system and NID panel for identification of Enterobacteriaceae, Vibrionaceae, and commonly isolated nonenteric gram-negative bacilli. J Clin Microbiol 2006; 44(3): 928-33.
- Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing. Seventeenth Informational Supplement. CLSI Document M100-S17, 2007. CLSI, Wayne, PA.
- Clinical and Laboratory Standards Institute. Methods for dilution antimicrobial susceptibility test for bacteria that grow aerobically. Approved Standard, 7th ed. CLSI Document M7-A7, 2006. CLSI, Wayne, PA.
- Begum D, Strockbine NA, Sowers EG, Jackson MP. Evaluation of a technique for identification of Shiga-like toxin-producing Escherichia coli by using polymerase chain reaction and digoxigenin-labeled probes. J Clin Microbiol 1993; 31(12): 3153-6.
- Lim KT, Yasin R, Yeo CC, Puthucheary S, Thong KL. Characterization of multidrug resistant ESBL-producing Escherichia coli isolates from hospitals in Malaysia. J Biomed Biotechnol 2009; 2009: 165637.
- Lévesque C, Piché L, Larose C, Roy PH. PCR mapping of integrons reveals several novel combinations of resistance genes. Antimicrob Agents Chemother 1995; 39(1): 185-91.
- White PA, McIver CJ, Rawlinson WD. Integrons and gene cassettes in the enterobacteriaceae. Antimicrob Agents Chemother 2001; 45(9): 2658-61.
- Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. 1989, 2nd ed. CSH Press, Cold Spring Harbor, New York.
- Looney WJ, Narita M, Mühlemann K. Stenotrophomonas maltophilia: an emerging opportunist human pathogen. Lancet Infect Dis 2009; 9(5): 312-23.
- Gales AC, Jones RN, Forward KR, Linares J, Sader HS, Verhoef J. Emerging importance of Acinetobacter species and Stenotrophomonas maltophilia as pathogens in severely ill patients: geographic patterns, epidemiological features, and trends in the SENTRY Antimicrobial Surveillance program (1997-1999). Clin Infect Dis 2001; 32(Suppl 2): S104-13.
- Falagas ME, Kastoris AC, Vouloumanou EK, Dimopoulos G. Community-acquired Stenotrophomonas maltophilia infections: a systematic review. Eur J Clin Microbiol Infect Dis 2009; 28(7): 719-30.
- Fedler KA, Biedenbach DJ, Jones RN. Assessment of pathogen frequency and resistance patterns among pediatric patient isolates: report from the 2004 SENTRY Antimicrobial Surveillance Program on 3 continents. Diagn Microbiol Infect Dis 2006; 56(4): 427-36.
- Mutlu M, Yılmaz G, Aslan Y, Bayramoğlu G. Risk factors and clinical characteristics of Stenotrophomonas maltophilia infections in neonates. J Microbiol Immunol Infect 2011; 44(6): 467-72.
- Caylan R, Kaklikkaya N, Aydin K, et al. An epidemiological analysis of Stenotrophomonas maltophilia strains in a university hospital. Jpn J Infect Dis 2004; 57(2): 37-40.
- Neela V, Rankouhi SZ, van Belkum A, Goering RV, Awang R. Stenotrophomonas maltophilia in Malaysia: molecular epidemiology and trimethoprim-sulfamethoxazole resistance. Int J Infect Dis 2012; 16(8): e603-7.
- Toleman MA, Bennett PM, Walsh TR. ISCR elements: novel gene-capturing systems of the 21st century? Microbiol Mol Biol Rev 2006; 70(2): 296-316.
- Köseoğlu O, Sener B, Gür D. Molecular epidemiology of Stenotrophomonas maltophilia strains isolated from paediatric patients. Mikrobiyol Bul 2004; 38(1-2): 9-19.
- Gülmez D, Çakar A, Şener B, Karakaya J, Hasçelik G. Comparison of different antimicrobial susceptibility testing methods for Stenotrophomonas maltophilia and results of synergy testing. J Infect Chemother 2010; 16(5): 322-8.
- Tatman-Otkun M, Gürcan S, Ozer B, Aydoslu B, Bukavaz S. The antimicrobial susceptibility of Stenotrophomonas maltophilia isolates using three different methods and their genetic relatedness. BMC Microbiol 2005; 5: 24.
- Song JH, Sung JY, Kwon KC, et al. Analysis of acquired resistance genes in Stenotrophomonas maltophilia. Korean J Lab Med 2010; 30(3): 295-300.
- Wu K, Wang F, Sun J, et al. Class 1 integron gene cassettes in multidrug-resistant gram-negative bacteria in southern China. Int J Antimicrob Agents 2012; 40(3): 264-7.
- Liaw SJ, Lee YL, Hsueh PR. Multidrug resistance in clinical isolates of Stenotrophomonas maltophilia: roles of integrons, efflux pumps, phosphoglucomutase (SpgM), and melanin and biofilm formation. Int J Antimicrob Agents 2010; 35(2): 126-30.
İletişim (Correspondence):
Uzm. Dr. Esra Özkaya,
Kahramanmaraş Necip Fazıl Şehir Hastanesi,
Mikrobiyoloji Laboratuvarı, Kahramanmaraş, Türkiye.
Tel (Phone): +90 344 228 2800/2159,
E-posta (E-mail): esragovce@hotmail.com