Doğu Karadeniz Bölgesi Hepatit C Hastalarında Hepatit C Virusu Genotiplerinin Belirlenmesi*
Determination of Hepatitis C Virus Genotypes Among Hepatitis C Patients in Eastern Black Sea Region, Turkey
Celal Kurtuluş BURUK, Gülçin BAYRAMOĞLU, Ahu REİS, Neşe KAKLIKKAYA, İlknur TOSUN, Faruk AYDIN
Karadeniz Teknik Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Trabzon.
Karadeniz Technical University Faculty of Medicine, Department of Medical Microbiology, Trabzon, Turkey.
* Bu çalışma, 6. Ulusal Tanısal ve Moleküler Mikrobiyoloji Kongresi (15-19 Haziran 2010, ODTÜ, Ankara)'nde sunulan bazı verileri kapsamaktadır.
ÖZET
Hepatit C virusu (HCV), transfüzyonla ilişkili kronik hepatitin majör etkeni olup, tüm dünyada olduğu gibi Türkiye'de de önemli bir halk sağlığı sorunudur. Altı majör genotipi ve çok sayıda yakın ilişkili alttipleri olan HCV'nin genotiplendirmesi; epidemiyolojik verilerin toplanması, antiviral tedavinin şekillendirilmesi ve prognozun belirlenmesinde büyük önem taşımaktadır. Bu çalışmada, Türkiye'nin Doğu Karadeniz Bölgesi illerinde yaşayan HCV-RNA pozitif bireylerde, HCV genotip dağılımının belirlenmesi amaçlanmıştır. Çalışmaya, Ocak 2009-Aralık 2012 tarihleri arasında Karadeniz Teknik Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı Hasta Hizmetleri Laboratuvarı Moleküler Mikrobiyoloji Birimine kabul edilen, HCV-RNA pozitif 304 hasta (151 erkek, 153 kadın; yaş aralığı: 11-93 yıl, yaş ortalaması: 55.2 ± 13.3 yıl) dahil edilmiştir. Hastalara ait plazma örneklerinde HCV genotipleri ticari yöntemler [INNO-LiPA HCV II (Innogenetics, Belçika) veya Abbott RealTime HCV Genotype II (Abbott Molecular Inc, ABD)] kullanılarak belirlenmiştir. Bu yöntemler ile bazı örneklerde ortaya çıkan şüpheli genotip sonuçları nedeniyle, çalışmada ayrıca genotipe özgül primerlerin kullanıldığı "in house" bir multipleks polimeraz zincir reaksiyonu (PCR) yöntemi de uygulanmıştır. Çalışmamızda, ülkemizin genel verilerine paralel olarak, Doğu Karadeniz Bölgesi'nde illere göre değişen oranlarda dört genotipin (1, 2, 3 ve 4) var olduğu; baskın genotipin 1 (%92.8) ve baskın alttipin 1b (%87.5) olduğu gösterilmiştir. HCV alttip 1a, genotip 2, 3 ve 4 prevalansı ise sırasıyla %5.3, %1.6, %4.9 ve %0.7 oranlarında belirlenmiştir. Ayrıca çalışmada, göreceli olarak daha az göç alan Giresun, Gümüşhane ve Bayburt'ta HCV genotip 1'in daha yüksek olduğu ve Trabzon, Rize ve Artvin'e ait verilerin yabancı uyruklu hasta yerleşiminden etkilendiği gözlenmiştir. Bu çalışma, Doğu Karadeniz Bölgesi illerinde yaşayan kronik hepatit C hastalarındaki HCV genotip dağılımı verilerinin sunulduğu ilk araştırmadır. Ayrıca bu çalışmada, HCV genotiplendirmesinde kullanılan ticari "line probe assay" (LiPA) testinde karşılaşılan "hayalet bant" ve gerçek zamanlı PCR testinde karşılaşılan genotip 4 içeren çoklu genotip saptanması gibi yöntemsel sorunların çözümünde, genotipe özgül multipleks PCR yönteminin Türkiye'de görülen genotipleri tanımlamada yararlı olabileceği kanısına varılmıştır.
Anahtar sözcükler: Hepatit C virusu; genotiplendirme; Doğu Karadeniz Bölgesi; Türkiye.
ABSTRACT
Hepatitis C virus (HCV), the major cause of transfusion-associated hepatitis, is an important public health problem in the world as well as in Turkey. HCV is grouped as six distinct genotypes and a large number of closely-related subtypes. Genotyping of HCV is an important tool for providing epidemiological data, prediction of prognosis, and optimization of antiviral therapy. This study was carried out to determine the distribution of HCV genotypes in hepatitis C patients residing in different provinces of the Eastern Black Sea Region, Turkey. A total of 304 HCV-RNA positive cases (151 male, 153 female; age range: 11-93 years, mean age: 55.2 ± 13.3 years) who were admitted to the Molecular Microbiology Unit of Department of Medical Microbiology, Karadeniz Technical University Faculty of Medicine, between January 2009 to December 2012, were included in the study. HCV genotypes were detected in plasma samples of the patients by using commercial assays [INNO-LiPA HCV II (Innogenetics, Belgium) or Abbott RealTime HCV Genotype II (Abbott Molecular Inc, USA)]. Due to the ambiguous genotyping results in some samples with these methods, an in-house multiplex polymerase chain reaction (PCR) assay with genotype-specific primers was also used in the study. Similar to the previous reports from Turkey, our results showed that four HCV genotypes (1, 2, 3, and 4) prevailed in the Eastern Black Sea Region and the predominant genotype and subtype were genotype 1 (92.8%) and 1b (87.5%), respectively. Distrubution of genotypes were observed to vary according to the province. Prevalences of subtype 1a, genotype 2, 3, and 4 were noted as 5.3%, 1.6%, 4.9%, and 0.7%, respectively. Furthermore, the samples from Giresun, Gumushane and Bayburt provinces, which are relatively less immigrated, had higher genotype 1, and the prevalence rates in the region was affected by the presence of non-citizen residents. This study is the first report on distribution of HCV genotypes in chronic hepatitis C patients living in the provinces of Eastern Black Sea Region. Moreover, genotype-specific multiplex PCR assay could be useful in resolving certain methodological problems such as "ghost bands" encountered in line probe assay (LiPA) and multiple genotypes (including genotype 4) observed in real-time PCR during the characterization of HCV genotypes seen in Turkey.
Key words: Hepatitis C virus; genotyping; Eastern Black Sea Region, Turkey.
Geliş Tarihi (Received): 29.04.2013 • Kabul Ediliş Tarihi (Accepted): 29.07.2013
GİRİŞ
Hepatit C virusu (HCV) 1989 yılında tanımlandıktan sonra posttransfüzyon ve sporadik non-A ve non-B hepatitlerinin en önemli etiyolojik ajanı olduğu görülmüştür1. HCV, Flaviviridae ailesinin Hepacivirus cinsine ait küçük, zarflı bir virustur2. Persistan enfeksiyon oranı %85'lere ulaşmakta ve kronik hepatit C, hepatik steatoz, siroz ve hepatoselüler karsinom ile ilişkilendirilmektedir. Dünya nüfusunun yaklaşık %3'ünün HCV ile enfekte olduğu tahmin edilmektedir. Türkiye'de görülme sıklığı %0.3-1.8 (ortalama %0.5) arasındadır3.
HCV %31-34 nükleotid, yaklaşık %30 aminoasit dizi farklılığı bulunan altı genotipe ve çok sayıda yakın ilişkili alttipe ayrılmaktadır2. HCV genotiplerinin dağılımında coğrafik farklılıklar rol oynamaktadır. Nitekim genotip 1a Amerika ve Kuzey Avrupa'da; genotip 1b tüm dünyada, özellikle Kuzey Amerika, Avrupa ve Japonya'da; genotip 2 Akdeniz ülkeleri ve Uzak Doğu'da; genotip 3 Avrupa'da; genotip 4 Ortadoğu'da; genotip 5 Güney Afrika'da; genotip 6 Hong Kong, Vietnam ve Avustralya'da baskın olarak görülmektedir4. Ülkemizde HCV genotip 1 baskın olmakla birlikte; genotip 2, 3 ve 4 de bölgelere göre değişen sıklıklarda görülmektedir5,6,7,8,9,10,11,12,13,14,15,16,17,18.
Hepatit C hastalarında antiviral tedavinin süresini ve sürdürülebilir virolojik yanıtı etkileyen faktörlerin başında genotip farklılıkları gelmektedir19,20,21. İnterferon tedavisine genotip 1 ve 4 daha dirençli iken, genotip 2 ve 3 daha iyi yanıt vermektedir4. Yeni geliştirilen ilaçlara yanıtta da genotipler arası farklılıkların olduğu gösterilmiştir22. HCV genotiplerinin belirlenmesi, klinik katkısının yanında epidemiyolojik veri sağlaması açısından de önem taşımaktadır3,23. HCV genotiplerinin belirlenmesinde altın standart yöntem dizi analizi olmakla birlikte; düşük maliyet, uygulama kolaylığı, daha az cihaz ve donanım gerektirmesi nedeniyle polimeraz zincir reaksiyonu (polymerase chain reaction, PCR) sonrası "restriction fragment length polymorphism (RFLP)", "line probe assay (LiPA)", genotipe özgül primerlerle yapılan PCR ve gerçek zamanlı PCR (real-time PCR, Rt-PCR) yöntemleri kullanılmaktadır24.
Bir coğrafi alandaki HCV genotip dağılımı, kültürel çeşitliliğin artışıyla değişebilmektedir25. Doğu Karadeniz Bölgesi, tarih boyunca Doğu ve Batı arasındaki önemli geçiş ve göç yollarından birisi olmuştur. Ayrıca, özellikle son yıllarda bölgedeki yabancı uyruklu insan yerleşimi artmaktadır. Bu durum, önemli bir halk sağlığı sorunu olan HCV enfeksiyonunun genotipik anlamda bölgede çeşitlenmiş olabileceğini düşündürmektedir. Bununla birlikte, literatürde Doğu Karadeniz Bölgesi'ndeki HCV genotip dağılımına ait kapsamlı bir bilgi bulunmamaktadır. Bu çalışmada Doğu Karadeniz Bölgesi illerinde yaşayan HCV-RNA pozitif bireylerde var olan HCV genotiplerinin dağılımlarının belirlenmesi amaçlanmıştır.
GEREÇ ve YÖNTEM
Çalışmada, Ocak 2009-Aralık 2012 tarihleri arasında, Doğu Karadeniz Bölgesi için referans konumda olan Karadeniz Teknik Üniversitesi Tıp Fakültesi, Farabi Hastanesi Tıbbi Mikrobiyoloji Anabilim Dalı, Hasta Hizmetleri Laboratuvarı Moleküler Mikrobiyoloji Birimine kabul edilen, farklı hastalara ait HCV-RNA pozitif plazma örnekleri genotipleri açısından analiz edildi. Çalışma popülasyonu, Doğu Karadeniz Bölgesi illeri olan Trabzon, Giresun, Rize, Artvin, Gümüşhane ve Bayburt'ta ikamet eden ve kronik hepatit C enfeksiyonu olan, 12'si yabancı uyruklu toplam 304 hastadan oluşmaktaydı. Çalışma, Karadeniz Teknik Üniversitesi Tıp Fakültesi Klinik Araştırmalar Etik Kurulu onayı (Protokol no: 2013/87) ile gerçekleştirildi.
Belirtilen tarihler arasında HCV-RNA pozitifliği iki farklı ticari Rt-PCR (COBAS® AmpliPrep/COBAS® TaqMan® HCV Test, Roche Diagnostics Corporation, ABD ve Abbott RealTime HCV Assay, Abbott Molecular Inc, ABD) testlerinden biriyle saptandı. HCV-RNA pozitif 304 örneğin 159'unda INNO-LiPA HCV II (Innogenetics, Belçika) ve 145'inde Abbott RealTime HCV Genotype II (Abbott Molecular Inc, ABD) testleri kullanılarak HCV genotipleri belirlendi. LiPA ve Rt-PCR yöntemlerine dayalı genotiplendirme testleri, kılavuzlarında belirtildiği şekilde uygulandı ve yorumlandı. LiPA yönteminde öncelikle cDNA sentezi yapıldı. Biyotin ile işaretli primerler kullanarak cDNA'dan elde edilen PCR ürünlerinin membran üzerine bağlı genotipe özgül HCV dizilerine hibridizasyonu enzimatik olarak gösterildi. Oluşan bantlar kılavuz ile karşılaştırılarak genotipe karar verildi. Rt-PCR yönteminde ise genotipe özgül floresan işaretli oligonükleotid primer ve problar içeren üç farklı multipleks reaksiyon karışımına doğrudan viral RNA eklendi. Rt-PCR sonunda oluşan amplifikasyon eğrilerinin yazılım tarafından (Abbott m2000rt) analiziyle genotip sonuçları elde edildi.
LiPA yöntemiyle çalışılan bazı örneklerde genotip 1b paternine ek ve başka herhangi bir paterne uymayan bantların görülmesi sonucu tiplendirilememeleri, ucuz ve kolay olan ikinci bir yöntemin laboratuvarda gerekliliğini gösterdi. Bu nedenle Ohno ve arkadaşları26 tarafından kullanılan genotipe özgül primerler (Tablo I) ve yöntem ile önce tüm örnekler; sonuçlardaki uyum belirlendikten sonra ise tanımlanmış paterne ek bant bulunduranlar ile 1b dışında sonuç saptanan örnekler yeniden test edildi. Aynı şekilde LiPA testlerine göre daha kolay ve hızlı olduğu için geçilen Rt-PCR yönteminde çoğul genotip ve 1b dışında sonuç saptanan örnekler yeniden test edildi.
Genotipe özgül multipleks PCR (mPCR) yönteminde kısaca, örneklerdeki HCV-RNA'ları EZ1 Virus Mini Kit v2.0 (Qiagen, Almanya) ve BioRobot EZ1 platformu (Qiagen, Almanya) kullanılarak izole edildikten sonra, "Reverse Transcription System" (Promega, ABD) ile komplementer DNA (cDNA) elde edildi. cDNA'lar kullanılarak Sc2 ve Ac2 primerleri ile birinci tur PCR; sonrasında multipleks 1 ve 2 master mikslerinde ikinci tur reaksiyon gerçekleştirildi. Ürünler %2'lik agaroz jelde yürütülerek UV ilüminatörde görüntülendi.
BULGULAR
Bu çalışmada, Doğu Karadeniz Bölgesi illerinde ikamet eden, 11-93 yaş arasındaki (ortalama yaş: 55.2 ± 13.3 yıl), 151 (%49.7) erkek ve 153 (%50.3) kadın toplam 304 hastaya ait kan örneklerinde HCV genotipleri belirlenmiştir. LiPA yönteminde genotip 1b paternine ek bant görülen iki örnek, genotipe özgül mPCR ile yeniden test edildiğinde M1 multipleks tüpünde genotip 1b ile uyumlu 234 baz çifti (bç) uzunluğunda ürün elde edilmiştir. Rt-PCR yöntemi ile çalışılan örneklerin üçünde genotip 1b ve genotip 4 ile ilişkili amplifikasyon eğrilerinin birlikte oluştuğu ve yazılım tarafından genotip 1b-4 şeklinde rapor edildiği izlenmiştir. Bu örnekler genotipe özgül mPCR ile yeniden test edildiğinde tamamında sadece genotip 1b ile uyumlu 234 bç uzunluğunda ürün oluştuğu belirlenmiştir.
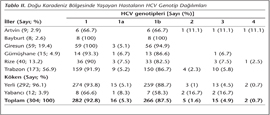
Doğu Karadeniz Bölgesi'nde yaşayan hepatit C hastalarındaki baskın virus genotipi 1 (%92.8) olarak saptanmış; hastaların %87.5'inin genotip 1b ile enfekte olduğu belirlenmiş, ancak genotip 2, 3 ve 4 enfeksiyonlarının da bulunduğu gözlenmiştir (Tablo II). Bölgede yerleşik bulunan 12 yabancı uyruklu hastada da, daha düşük oranda (%58.3) olmakla birlikte genotip 1b'nin daha sık olduğu tespit edilmiştir. Tamamı eski Sovyetler Birliği'nden ayrılan ülkelerden gelen yabancı uyruklu hastalarda genotip 4'e rastlanmamıştır (Tablo II).
TARTIŞMA
HCV genotiplerinin bilinmesi; epidemiyolojik verilerin toplanması, aşı geliştirme çalışmaları, tedavinin şekillendirilmesi ve prognozun belirlenmesinde önemlidir4,25. Bu amaçla ticari veya "in house" validasyonu yapılmış dizi analizi, PCR-RFLP, ters hibridizasyon LiPA, genotipe özgül PCR ve Rt-PCR kullanılmaktadır24,26. Ancak altın standart yöntem olarak kullanılan dizi analizi dahil tüm yöntemlerde genotip tanımlamada bazı zorluklarla karşılaşılmaktadır24,27,28,29,30. Çalışmamızda, LiPA kullanılarak yapılan HCV genotiplendirme sürecinde iki örnekte genotip 1b için tanımlanan bantlara ek bantlar ile karşılaşılmıştır. "Hayalet bant" olarak adlandırılan bu bantlar sonucu etkileyebilmektedir24. Rt-PCR yöntemiyle yapılan genotiplendirmede de üç örnekte genotip 1 ve genotip 4 birlikteliği görülmüştür. Yapılan çalışmalarda, Rt-PCR yöntemiyle çalışılan Abbott HCV genotiplendirme testlerinde kullanılan genotip 4 probunun özgüllüğünün sorunlu olduğu24 ve çoklu genotip saptanan (genellikle genotip 1 ve 4 birlikteliği) örneklerde genotipin 1 olduğu rapor edilmiştir28. Laboratuvarda ikinci bir yöntemin kullanılması duyarlılığı artırmaktadır27. Bu çalışmada, standardize ticari LiPA ve Rt-PCR yöntemlerinde bazı örneklerde ortaya çıkan genotiplendirememe sorunu, düşük maliyetli ikinci bir yöntem olan Ohno ve arkadaşlarının26 genotipe özgül multipleks PCR yöntemiyle çözülmüştür.
HCV genotiplerinin dağılımında coğrafi farklılıklar rol oynamaktadır. Yayımlanan çalışmalarda ülkemizdeki genotip dağılımlarına bakıldığında genotip 1b'nin (%52.8-100) baskın olduğu görülmektedir. Genel olarak iç bölgelerde (Kayseri hariç) genotip 1b oranı yüksek; büyük şehirler ve göç alan illerde daha düşük olarak rapor edilmektedir5,6,7,8,9,10,11,12,13,14,15,16,17,18. Genotip 1b için en düşük oran (%52.8-57.6) Kayseri'den bildirilmiştir14,17. Çalışmamızda, Bayburt ili hastalarının tamamının HCV genotip 1b ile enfekte olduğu, Giresun'da da genotip 1b'nin diğer illere göre daha yüksek oranda bulunduğu görülmüştür. Bölgede en düşük oran %66.7 ile Artvin örneklerinde belirlenmiştir. Genotip 1a ise İzmir, Ankara, Afyon ve Diyarbakır illerinde daha yüksek olarak (%19.1-22.7) rapor edilmektedir5,7,11,16. Diğer çalışmalarda oranlar %0-10 arasında değişmektedir6,9,12. Bu çalışmaya konu illerde de benzer şekilde oran %0-7.5 arasında bulunmuştur.
Genotip 2 prevalansı bölgesel farklılıklar ile birlikte %0-3.8 olarak bildirilmektedir7,8,9,11. Çalışmamızda HCV genotip 2, Giresun, Bayburt, Gümüşhane ve Rize örneklerinde saptanmamış; Trabzon örneklerinde %2.3, Artvin örneklerinde ise %11.1 oranında tespit edilmiştir (Tablo II). HCV genotip 3 prevalansı dikkate alındığında, ülkemiz genelinde oranların düşük (%0-5.4) olduğu5,6,7,16 izlenmekte; ancak Küçüköztaş ve arkadaşlarının12 %9.6 oranında pozitiflik bildirdiği görülmektedir. Bizim çalışmamızda Bayburt ve Giresun örneklerinde genotip 3 saptanmazken, Trabzon için bu oran %5.8, Rize için %7.5 olarak belirlenmiştir. Gümüşhane (1/15; %6.7) ve Artvin (1/9; %11.1) örneklerinin ise birer tanesinde genotip 3 belirlenmesine karşın, pozitif olgulardan birinin Gümüşhane'ye tayin olmuş bir devlet memuru, diğerinin de Artvin'de yaşayan yabancı uyruklu bir olgu olduğu saptanmıştır. Daha ziyade Ortadoğu ülkelerinde görülen HCV genotip 4, ülkemizde Kayseri ve Afyon illerinde yüksek prevalansa sahiptir4,7,14,17. Çalışmamızda Artvin ve Rize hastalarına ait birer örnekte (sırasıyla %11.1 ve %2.5) genotip 4 saptanmıştır.
Bölgesel bağlamda düşünüldüğünde, Doğu Karadeniz Bölgesi'nde yaşayan hepatit C hastalarındaki baskın HCV genotipinin Türkiye verileri ile paralel bir şekilde genotip 1b (%87.5) olduğu saptanmıştır. Diğer genotiplerden genotip 1a (%5.3) ve genotip 3 (%4.9) oranının birbirine yakın olduğu görülmüş, genotip 2 (%1.6) ve genotip 4 (%0.7) daha az oranda saptanan tipler olmuştur.
Son zamanlarda özellikle Doğu Karadeniz Bölgesi'nde yaşayan yabancı uyruklu insan sayısı artmaktadır. Bu çalışmada incelenen örneklerden 12 (%3.9)'sinin yerleşik yaşayan yabancı uyruklu HCV hastalarına ait olduğu görülmüştür. Bu hastalara ait veriler irdelendiğinde genotip 1b'nin baskın olduğu (%58.3), genotip 2 ve 3'ün Türkiye ortalamalarının üzerinde prevalansa sahip olduğu (her ikisi de %16.7) belirlenmiştir. Bu veri, ilerleyen dönemde genotip 2 ve genotip 3 oranlarının bölgede artabileceğini düşündürmektedir.
Sonuç olarak, bu çalışmada sunulan Doğu Karadeniz Bölgesi illerinde yaşayan kronik hepatit C hastalarında HCV genotip dağılımı verileri ilk olma niteliği taşımaktadır. Çalışma, bölgeye ait prevalans verilerinin Türkiye geneli ile benzer olmakla birlikte; göreceli olarak daha az göç alan Giresun, Gümüşhane ve Bayburt'ta HCV genotip 1'in daha yüksek olduğunu ve verilerin, kültürel çeşitliliğin artışından etkilendiğini göstermektedir. Ayrıca bu çalışmada, HCV genotiplendirmede karşılaşılan yöntemsel sorunların çözümünde genotipe özgül multipleks PCR'nin Türkiye'de görülen genotipleri tanımlamada ticari yöntemlere ikincil test olarak katkı sağlayabileceği düşünülmektedir.
KAYNAKLAR
- Kuo G, Choo QL, Alter HJ, et al. An assay for circulating antibodies to a major etiologic virus of human non-A, non-B hepatitis. Science Apr 1989; 244(4902): 362-4.
- Simmonds P, Bukh J, Combet C, et al. Consensus proposals for a unified system of nomenclature of hepatitis C virus genotypes. Hepatology 2005; 42(4): 962-73.
- Sünbül M. HCV enfeksiyonunun epidemiyolojisi ve korunma, s: 208-19. Tabak F, Tekeli E, Balık İ (ed), Viral Hepatit 2007. 2007, 1. baskı. Deniz Ofset, İstanbul.
- Hnatyszyn HJ. Chronic hepatitis C and genotyping: the clinical significance of determining HCV genotypes. Antivir Ther 2005; 10(1): 1-11.
- Abacıoglu YH, Davidson F, Tuncer S, et al. The distribution of hepatitis C virus genotypes in Turkish patients. J Viral Hepat 1995; 2(6): 297-301.
- Özaçar T, Altuglu I, Zeytinoglu A ve ark. Kronik C hepatitinde HCV genotiplerinin dağılımı. Mikrobiyol Bul 2001; 35 (3): 451-8.
- Kalayci R, Altindiş M, Gülamber G, Demirtürk N, Akcan Y, Demirdal T. Kronik hepatit B ve hepatit C'li hastalarda genotip dağılımı ve hepatit B olgularında direnç paterninin araştırılması. Mikrobiyol Bul 2010; 44(2): 237-43.
- Şanlıdağ T, Akçalı S, Özbakkaloğlu B, Ertekin D, Akduman E. Manisa bölgesinde hepatit C virus genotiplerinin dağılımı. Mikrobiyol Bul 2009; 43(4): 613-8.
- Küçüköztaş MF, Özgüneş N, Yazıcı S. Kronik hepatit C'li hastalarda hepatit C virusu (HCV) genotipleri ile alanin aminotransferaz ve HCV-RNA düzeyleri arasındaki ilişkinin araştırılması. Mikrobiyol Bul 2010; 44(1): 111-5.
- Aktaş E, Ogedey ED, Külah C, Beğendik Cömert F. Zonguldak bölgesinde hepatit C virusu genotipleri. Mikrobiyol Bul 2010; 44(4): 647-50.
- Bozdayı G, Verdi H, Rota S ve ark. Hemodiyaliz hastalarında hepatit C virus enfeksiyon varlığının araştırılması ve HCV genotip dağılımının belirlenmesi. Mikrobiyol Bul 2002; 36 (3): 291-304.
- Ural O, Arslan U, Fındık D. Konya bölgesinde hepatit C virusu genotip dağılımı. İnfeksiyon Dergisi 2007; 21(4): 175-81.
- Çelik C, Bakıcı MZ, Kaygusuz R, Ertürk R. Sivas yöresindeki HCV genotip dağılımlarının araştırılması. Viral Hepatit Derg 2010; 16(3): 106-10.
- Gökahmetoğlu S, Atalay MA, Kılınç A. Hepatit C virüs genotiplerinin pirosekanslama yöntemi ile belirlenmesi. Erciyes Tıp Derg 2011; 33(2): 99-102.
- Yarkın F, Hafta A. Kronik hepatit C enfeksiyonu olan hastalarda hepatit C virüs (HCV) genotiplerinin dağılımı. Viral Hepatit Derg 2000; 6(3): 164-8.
- Çil T, Özekinci T, Göral V, Altıntaş A. Güneydoğu Anadolu Bölgesi'nde hepatit C virüsü genotipleri. Turkiye Klinikleri J Med Sci 2007; 27(4): 496-500.
- Kayman T, Karakükçü Ç, Karaman A, Gözütok F. Kayseri bölgesinde hepatit C virüs enfeksiyonunun genotip dağılımı. Türk Mikrobiyol Cem Derg 2012; 42(1): 21-6.
- Tezcan S, Ulger M, Aslan G ve ark. Mersin ilinde hepatit C virusu genotip dağılımının belirlenmesi. Mikrobiyol Bul 2013;47(2):332-8.
- EASL International Consensus Conference on Hepatitis C. Paris, 26-28 February 1999, Consensus Statement. European Association for the Study of the Liver. J Hepatol 1999; 30(5): 956-61.
- Mangia A, Ricci GL, Persico M, et al. A randomized controlled trial of pegylated interferon alpha-2a (40 KD) or interferon alpha-2a plus ribavirin and amantadine vs interferon alpha-2a and ribavirin in treatment-naïve patients with chronic hepatitis C. J Viral Hepat 2005;12(3): 292-9.
- Kim SR, El-Shamy A, Imoto S, et al. Prediction of response to pegylated interferon/ribavirin combination therapy for chronic hepatitis C genotype 1b and high viral load. J Gastroenterol 2012; 47(10): 1143-51.
- Foster GR, Hézode C, Bronowicki JP, et al. Telaprevir alone or with peginterferon and ribavirin reduces HCV RNA in patients with chronic genotype 2 but not genotype 3 infections. Gastroenterology 2011; 141(3): 881-9.
- Cantaloube JF, Gallian P, Attoui H, Biagini P, De Micco P, de Lamballerie X. Genotype distribution and molecular epidemiology of hepatitis C virus in blood donors from southeast France. J Clin Microbiol 2005; 43(8): 3624-9.
- Weck K. Molecular methods of hepatitis C genotyping. Expert Rev Mol Diagn 2005; 5(4): 507-20.
- Ghany MG, Strader DB, Thomas DL, Seeff LB. Diagnosis, management, and treatment of hepatitis C: an update. Hepatology 2009; 49(4): 1335-74.
- Ohno O, Mizokami M, Wu RR, et al. New hepatitis C virus (HCV) genotyping system that allows for identification of HCV genotypes 1a, 1b, 2a, 2b, 3a, 3b, 4, 5a, and 6a. J Clin Microbiol 1997; 35(1): 201-7.
- Furione M, Simoncini L, Gatti M, et al. HCV genotyping by three methods: analysis of discordant results based on sequencing. J Clin Virol 1999; 13(3): 121-30.
- Ciotti M, Marcuccilli F, Guenci T, et al. A multicenter evaluation of the Abbott RealTime HCV Genotype II assay. J Virol Methods 2010; 167(2): 205-7.
- Schutzbank TE, Sefers SE, Kahmann N, Li H, Tang YW. Comparative evaluation of three commercially available methodologies for hepatitis C virus genotyping. J Clin Microbiol 2006; 44(10): 3797-8.
- Nolte FS, Green AM, Fiebelkorn KR, et al. Clinical evaluation of two methods for genotyping hepatitis C virus based on analysis of the 5' noncoding region. J Clin Microbiol 2003; 41(4): 1558-64.
İletişim (Correspondence):
Yrd. Doç. Dr. Celal Kurtuluş Buruk,
Karadeniz Teknik Üniversitesi Tıp Fakültesi,
Tıbbi Mikrobiyoloji Anabilim Dalı,
61000 Trabzon, Türkiye.
Tel (Phone): +90 462 377 5404,
E-posta (E-mail): kburuk@ktu.edu.tr