Candida albicans Suşlarının
Genotiplendirmesinde 25S İntron Analizini Takiben
Restriksiyon Enzim Analizi Uygulanması*
25S
Intron Analysis Followed by Restriction Enzyme Digestion Performed for
Genotyping
Candida albicans Isolates
Zeynep
Ceren KARAHAN1, Begüm SARAN1, Sevinç YENİCE1,
Handan AĞIRBAŞLI2, Özay ARIKAN AKAN3,
Alper TEKELİ1
1 Ankara Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Ankara.
1 Ankara University Faculty of Medicine, Department of Medical Microbiology, Ankara, Turkey.
2 İstanbul Üniversitesi İstanbul Tıp Fakültesi, Mikrobiyoloji Laboratuvarı, İstanbul.
2 Istanbul University Istanbul Faculty of Medicine, Microbiology Laboratory, Istanbul, Turkey.
3 Ankara Üniversitesi Tıp Fakültesi, İbn-i Sina Hastanesi, Merkez Laboratuvarı, Ankara.
3 Ankara University Faculty of Medicine, Ibn-i Sina Hospital, Central Laboratory, Ankara, Turkey.
* Bu araştırma, Ankara Üniversitesi Biyoteknoloji Enstitüsü desteği ile (Proje no. 2001K120240) gerçekleştirilmiş ve çalışmanın bir bölümü, 18. Avrupa Klinik Mikrobiyoloji ve Enfeksiyon Hastalıkları Kongresi (19-22 Nisan 2008, Barcelona, İspanya)'nde poster olarak sunulmuştur.
ÖZET
Candida albicans, özellikle bağışıklığı baskılanmış konaklarda enfeksiyon etkeni olarak en sık izole edilen fungal patojendir. Uygun enfeksiyon kontrol stratejilerinin geliştirilmesi ve epidemiyolojik veri toplanması açısından, enfeksiyon etkeni olan mikroorganizmaların genotiplendirilmesi önem taşımaktadır. 25S intron analizi, C.albicans suşlarının genotiplendirilmesinde kullanılan kolay ve güvenilir bir yöntem olarak kabul edilmektedir. Bununla birlikte, ayırım gücünün düşük olması, epidemiyolojik araştırmalarda bu yöntemin kullanımını sınırlandırmaktadır. Bu çalışmada, enfeksiyon etkeni olarak izole edilen C.albicans suşlarının polimeraz zincir reaksiyonu (PCR) ile 25S intron analizini takiben, elde edilen PCR ürünlerinin restriksiyon enzim analizine tabi tutulmasıyla ayırım gücü daha yüksek olan bir genotiplendirme sistemi oluşturulması amaçlanmıştır. Bu çalışmaya, çeşitli klinik örneklerden (121 kan, 69 balgam, 36 vajinal akıntı, 26 yara, 8 idrar) enfeksiyon etkeni olarak izole edilmiş 260 adet C.albicans suşu dahil edilmiştir. Tüm izolatlara, PCR ile literatürde belirtildiği şekilde 25S intron analizi uygulanmış ve elde edilen PCR ürünleri HaeIII restriksiyon enzimi ile kesilerek genotiplendirilmiştir. Her iki yöntemin ayırım gücü ayrı ayrı hesaplanmıştır. 25S intron analizi ile 184 (%70.8) izolat genotip A, 42 (%16.2) izolat genotip B ve 34 (%13) izolat da genotip C olarak bulunmuş; yöntemin ayırım gücü 0.46 olarak hesaplanmıştır. PCR ürünlerinin HaeIII ile kesimi sonucunda genotip A'da 10, genotip B'de bir ve genotip C'de beş ayrı kesim paterni (genotip) tespit edilmiş; böylece restriksiyon enzim analizi eklenmesiyle elde edilen genotip sayısı 16'ya, yöntemin ayırım gücü de 0.79'a yükselmiştir. Farklı genotiplendirme yöntemlerinin birlikte kullanımı, genotip sayısını artırarak ayırım gücünü yükseltmekle birlikte, aynı suşların farklı genotiplere dağılmasıyla sonuçlanabilmekte ve bu durum değerlendirmede güçlüklere neden olabilmektedir. 25S intron analizini takiben HaeIII restriksiyon enzim analizi uygulanması ise, tamamen farklı bir genotiplendirme yöntemi eklenmesine gerek kalmaksızın yöntemin ayırım gücünü yükseltmekte ve yöntemi epidemiyolojik araştırmalar ve klinik izolatların genotiplendirilmesi için daha uygun hale getirmektedir. HaeIII yerine başka enzimlerin kullanılması yöntemin ayırım gücünü daha da yükseltebilir. Bu yöntemle elde edilen genotiplerle hasta özellikleri, klinik veriler, antifungal duyarlılıklar gibi parametreler arasında ilişki olup olmadığını araştırmak için daha detaylı araştırmalar planlanabilir.
Anahtar sözcükler: Candida albicans; genotip; polimeraz zincir reaksiyonu; PCR; 25S intron analizi; restriksiyon enzim analizi.
ABSTRACT
Candida albicans is the most frequently encountered fungal pathogen especially in the immunocompromised hosts. Genotyping clinical microbial isolates is important for obtaining epidemiological data and for establishing appropriate infection control strategies in the hospital setting. 25S intron analysis is an easy and reliable method used for genotyping C.albicans strains. As it has a low discriminatory power, its use is limited in epidemiological studies. In this study, our aim was to genotype clinical C.albicans isolates by using 25S intron analysis followed by restriction enzyme digestion in order to develop a more discriminative genotyping system for C.albicans. A total of 260 clinical C.albicans strains isolated from various infection sites (121 blood, 69 sputum, 36 vaginal discharge, 26 wound, 8 urine samples) were genotyped by 25S intron analysis, and all the products obtained by polymerase chain reaction (PCR) were digested with HaeIII restriction enzyme. Discriminatory power of each method was calculated. Among the isolates 184 (70.8%) were classified as genotype A, 42 (16.2%) as genotype B, and 34 (13%) as genotype C by 25S intron analysis. Discriminatory power of the method was calculated as 0.46. HaeIII restriction of genotype A, B and C isolates produced ten, one, and five restriction patterns (genotypes), respectively. By the addition of restriction enzyme analysis, the number of genotypes obtained was increased to 16, and the discriminatory power of the method to 0.79. Combining different genotyping methods increases the discriminatory power by increasing the number of genotypes obtained. However, there is also a risk to split certain strains in different genotypes by the different methods used and this makes the genotypic evaluation more difficult. On the other hand, combining 25S intron analysis with restriction enzyme analysis increases the discriminatory power without introducing a totally different method, and makes the method more suitable for epidemiological purposes and for genotyping clinical isolates. Different enzymes instead of HaeIII should be tested to evaluate the effect on the discriminatory power. In order to evaluate the relationship between the genotypes obtained by this method and parameters such as patient characteristics, clinical data, and antifungal susceptibilities, more sophisticated studies can be performed.
Key words: Candida albicans; genotype; polymerase chain reaction; PCR; 25S intron analysis; restriction mapping.
Geliş Tarihi (Received): 11.08.2011 • Kabul Ediliş Tarihi (Accepted): 07.01.2012
GİRİŞ
Candida albicans, insanlarda normal cilt ve mukozaların flora elemanı olarak kabul edilen, ancak bağışıklık sistemi baskılanmış konaklarda ağır sistemik enfeksiyonlar oluşturabilen, fırsatçı bir maya mantarıdır. Candida türünün en patojen üyesi olarak, sistemik mantar enfeksiyonlarının %50-70'inden sorumlu tutulmaktadır1,2,3. Kandidemi mortalitesi, altta yatan hastalığın ciddiyetine bağlı olarak %30-40 arasında değişmektedir4.
Uygun enfeksiyon kontrol stratejilerinin geliştirilmesi ve epidemiyolojik veri toplanması açısından, enfeksiyon etkenlerinin genotiplendirilmesi önemlidir. Moleküler yöntemlerin gelişmesi ve polimeraz zincir reaksiyonu (PCR)'nun keşfi sonrasında, diğer etkenler için olduğu gibi, Candida türlerinin genotiplendirilmesinde de yeni moleküler yöntemler kullanılmaya başlanmıştır. Bu yöntemler arasında, genom dizileme, multilokus dizi tiplendirmesi (MLST), multilokus enzim elektroforezi (MLEE), restriksiyon enzim analizi (REA), "pulsed field" jel elektroforezi (PFGE), mikrosatellit analizi ve "randomly amplified polymorphic DNA" analizi (RAPD) sayılabilir5,6,7,8,9,10,11,12,13,14. Bu yöntemler içerisinde, suşların genetik yakınlığı hakkında en doğru sonuç vereni ve en güvenilir olanı dizi analizi olup, Candida suşlarının genotiplendirilmesinde altın standart olarak kabul edilmektedir13,14. Bununla birlikte, yöntemin pahalı olması, özel donanım ve deneyim gerektirmesi nedeniyle, her klinik örnek için uygulanması mümkün değildir.
Ribozomal diziler, birçok fungal patojenin genotiplendirilmesinde yaygın olarak kullanılan hedeflerdendir. McCullough ve arkadaşlarının13, 25S rRNA geni üzerinde aktarılabilir bir grup I intron varlığını göstermeye yönelik olarak geliştirdikleri genotiplendirme sistemi olan 25S intron analizinde, PCR ile elde edilen ürünün büyüklüğüne göre C.albicans suşları üç genotipe (Genotip A-C) ayrılmaktadır. Daha önce yaptığımız bir çalışmada, genotip A izolatları arasında restriksiyon enzim analiziyle gösterilebildiğini bulduğumuz ve dizi analiziyle doğruladığımız dizi değişkenliklerinin varlığı, 25S intron analizini takiben elde edilen ürünlerin enzimle kesilmesinin, genotip sayısını artırarak daha yüksek ayırım gücüne sahip, dolayısıyla epidemiyolojik araştırmalarda kullanmaya daha elverişli bir genotiplendirme sistemi sağlayabileceğini düşündürmüştür15. Bu çalışmanın amacı, klinik C.albicans izolatlarının 25S intron analizini takiben restriksiyon enzim analiziyle genotiplendirilmesidir.
GEREÇ ve YÖNTEM
C.albicans Suşları ve DNA Ekstraksiyonu
Çalışmaya, Haziran 1999-Mart 2009 tarihleri arasında Ankara Üniversitesi Tıp Fakültesi ve İstanbul Üniversitesi Tıp Fakültesi hastanelerinde ayaktan ya da yatarak tedavi gören hastalardan enfeksiyon etkeni olarak izole edilen 260 C.albicans izolatı dahil edildi. İzolatlar farklı hastalardan, farklı enfeksiyon alanlarından (121 kan, 69 balgam, 36 vajinal akıntı, 26 yara, 8 idrar örneği) izole edildi ve çalışılıncaya kadar %20 gliserol içeren beyin-kalp infüzyon besiyeri içerisinde -20°C'de saklandı. C.albicans tiplendirmesi rutin mikrobiyolojik yöntemlerle (Gram boyama ve germ-tüp testleri) ve API 20C AUX (BioMérieux, Marcy l'Etoile, Fransa) sistemi kullanılarak yapıldı.
Suşlardan DNA ekstraksiyonunda Kaiser ve arkadaşlarının16 önerdiği yöntem kullanıldı. Elde edilen DNA örnekleri çalışılıncaya kadar -20°C'de saklandı.
25S İntron Analizi ve Restriksiyon Enzim Analizi (REA)
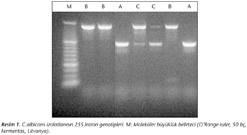
25S intron genotipleri, CA-INT-L ve CA-INT-F primerleri kullanılarak, literatürde belirtildiği şekilde PCR yöntemiyle gerçekleştirildi8,13. 450 baz çift (bç)'lik tek bant gözlenen izolatlar genotip A, 840 bç'lik tek bant gözlenen izolatlar genotip B, 450 ve 840 bç'lik iki bant veren izolatlar ise genotip C olarak tanımlandı (Resim 1).
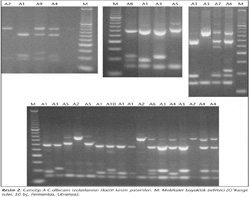
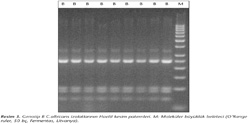
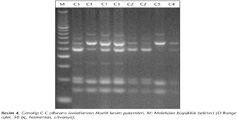
Tüm PCR ürünleri, uygun tampon içerisinde, 10 U HaeIII restriksiyon enzimi (Fermentas, Litvanya) kullanılarak 37°C'de bir gece tutularak kesildi. Ürünler %3'lük (w/v) agaroz jel elektroforezine (100V altında dört saat) tabi tutulduktan sonra etidyum bromür ile boyandı ve ultraviyole altında görüntülendi (Resim 2, 3, 4).
25S intron analizi ve REA için ayırım gücü (AG), aşağıdaki formüle göre hesaplandı:
Bu formülde s, elde edilen genotip sayısını; xj ve j genotipte yer alan izolatların sayısını; N çalışılan toplam örnek sayısını ifade etmektedir17.
Tüm PCR-REA uygulamaları, ikişer kere tekrarlanarak yöntemin tekrarlanabilirliği değerlendirildi.
BULGULAR
25S intron analizi ile, 260 izolatın 184 (%70.8)'ü genotip A, 42 (%16.2)'si genotip B ve 34 (%13)'ü genotip C olarak bulunmuş; yöntemin ayırım gücü 0.46 olarak hesaplanmıştır. PCR ürünlerinin HaeIII enzimi ile kesimi sonrasında genotip A, B ve C'de yer alan izolatlar sırasıyla 10 (A1-A10), 1 (B1) ve 5 (C1-C5) farklı kesim paterni oluşturmuştur. REA sonucu elde edilen A1 (n= 100), A2 (n= 42) ve A3 (n= 20) genotipleri, genotip A suşlarının %88'ini oluşturmakta olup, bu genotipler daha önceki çalışmamızda5 tespit ettiğimiz a, b, c, d ve e alt tiplerine karşılık gelmektedir. Tüm genotip B izolatları tek bir kesim paterni oluşturmuştur. HaeIII REA ile elde edilen bant sayısı 2-7 arasında, bant büyüklükleri yaklaşık 70-390 bç arasında değişmiştir. Bir genotipe düşen minimum ve maksimum izolat sayıları 1 ve 100 olmuştur (Tablo I). HaeIII REA yönteminin ayırım gücü 0.79 olarak hesaplanmıştır.
REA'nın tekrarlanması sonucunda, izolatlarda aynı bant paternleri tespit edilmiş, yöntemin tekrarlanabilir olduğu sonucuna varılmıştır.
TARTIŞMA
Enfeksiyon etkenlerinin genotiplendirmesi için bir moleküler yöntem seçilirken birkaç kriterin dikkate alınması gereklidir. Yöntem yeterli sayıda suşu birbirinden ayırt edebilmeli; yakın ilişkili veya birbirine uzak türleri tanımlayabilmeli; hızlı, güvenilir ve tekrarlanabilir sonuçlar vermelidir. Klinik mikrobiyoloji laboratuvarlarında yaygın olarak kullanılabilmesi için uygulaması kolay olmalı ve özel donanım gerektirmemelidir1,13,14,15. Bu çalışmada C.albicans suşlarının genotiplendirilmesinde kullandığımız 25S intron analizi kolay ve tekrarlanabilir bir yöntemdir. Değerlendirilen izolatlar arasında genotip A, diğer çalışmalarda olduğu gibi, suşların çoğunluğunu oluşturmuştur8,18,19.
Epidemiyolojik çalışmalarda kullanılabilmesi için bir genotiplendirme yönteminin ayırım gücünün 0.90'ın üzerinde olması arzu edilmektedir. Bu değerden uzaklaştıkça yöntemin güvenilirliği azalmaktadır. 25S intron analizi, C.albicans suşlarını sadece üç genotipe ayırdığından ve genetik olarak birbiriyle çok yakın olmayan türleri de aynı genotip altında topladığından, ayırım gücü istenen değerin çok altında kalmakta ve epidemiyolojik amaçlarla kullanılmasını engellemektedir18,20. 25S intron analizi sonrasında izolatlara HaeIII restriksiyon enzim analizinin uygulanması, elde edilen genotip sayısını 16'ya, ayırım gücünü ise 0.79'a yükseltmiştir. Hattori ve arkadaşları18 ile Iwata ve arkadaşları20, 25S intron analizinin ayırım gücünü artırmak için, yöntemi, tekrarlayan dizilere dayalı genotiplendirme [Repetitive sequence-based genotyping (RPS)] yöntemiyle kombine etmişler ve yöntemin ayırım gücünü sırasıyla 0.40'tan 0.67'ye ve 0.61'den 0.80'e yükseltmişlerdir. Bir genotiplendirme yönteminin bir başka yöntemle birlikte kullanılması, genotip sayısını artırarak yöntemin ayırım gücünü yükseltmektedir. Birlikte kullanılan yöntemler birbirinden tamamen farklı olabileceği gibi, belli bir yöntemin modifikasyonları (RAPD analizinde farklı primerler kullanılması veya mikrosatellit analizinde farklı bölgelerin hedeflenmesi gibi) da olabilir6,9,21,22. Birbirinden farklı genotiplendirme yöntemlerinin ayırım gücünü artırmak amacıyla kullanılmasının en önemli dezavantajı, bazı suşların ayrı genotiplerde kümelenebilmesi ve değerlendirmeyi zorlaştırmasıdır23,24,25. Bizim kullandığımız yöntemde suşlar, aynı genotip içerisinde alt tiplere ayrıldığından, farklı genotiplerde kümelenme riski ortadan kalkmaktadır.
Sonuç olarak; 25S intron analizini takiben REA analizi uygulanması, başka bir yöntem eklenmesine gerek kalmadan ayırım gücünü yükseltmekte ve epidemiyolojik çalışmalar için istenen değere yaklaştırmaktadır. Yöntem ucuz ve tekrarlanabilir bulunmuştur; uygulanması ve değerlendirmesi kolaydır; ek donanım gerektirmeden PCR imkanları olan laboratuvarlarda uygulanma şansı vardır. Farklı enzimlerin kullanımı ve farklı enzimlerle elde edilen sonuçların kombinasyonu, genotip sayısını daha da artırarak ayrım gücünü daha da yükseltebilir. Bu yöntemle elde edilen genotiplerle hasta özellikleri, klinik veriler, antifungal duyarlılıklar gibi parametreler arasında ilişki olup olmadığını araştırmak için daha detaylı araştırmalar planlanabilir.
KAYNAKLAR
- Dalle F, Franco N, Lopez J, et al. Comparative genotyping of Candida albicans bloodstream and non bloodstream isolates at a polymorphic microsattelite locus. J Clin Microbiol 2000; 38(12): 4554-9. [Özet] [Tam Metin] [PDF]
- Shi WM, Mei XY, Gao F, et al. Analyses of genital Candida albicans infection by rapid microsatellite markers genotyping. Chin Med J 2007; 120(11): 975-80. [Özet] [Tam Metin] [PDF]
- Tay ST, Chai HC, Na SL, Ng KP. Molecular subtyping of clinical isolates of Candida albicans and identification of Candida dubliniensis in Malaysia. Mycopathologia 2005; 159(3): 325-9. [Özet]
- Arendrup MC. Epidemiology of invasive candidiasis. Curr Opin Crit Care 2010; 16(5): 445-52. [Özet]
- Chen KW, Lo HJ, Lin YH, Li SY. Comparison of four molecular typing methods to assess genetic relatedness of Candida albicans clinical isolates in Taiwan. J Med Microbiol 2005; 54(Pt3): 249-58. [Özet] [Tam Metin] [PDF]
-
Clemons KV, Feroze F, Holmberg K, Stevens DA. Comparative analysis of
genetic variability among Candida albicans isolates from different
geographic locales by three genotypic methods. J Clin Microbiol 1997; 35(6):
1332-6.
[Özet] [PDF] - Dassanayake RS, Samaranayake LP. Amplification-based nucleic acid scanning techniques to assess genetic polymorphism in Candida. Crit Rev Microbiol 2003; 29(1): 1-24. [Özet]
- Karahan ZC, Güriz H, Ağırbaşlı H, et al. Genotype distribution of Candida albicans isolates by 25S intron analyses with regard to invasiveness. Mycoses 2004; 47(11-12): 465-9. [Özet]
- Odds FC, Brown AJ, Gow NA. Candida albicans genome sequence: a platform for genomics in the absence of genetics. Genome Biol 2004; 5(7): 230. [Özet] [Tam Metin] [PDF]
- Odds FC, Jacobsen MD. Multilocus sequence typing of pathogenic Candida species. Eukaryot Cell 2008; 7(7): 1075-84. [Tam Metin] [PDF]
- Pujol C, Joly S, Lockhart SR, Noel S, Tibayrenc M, Soll DR. Parity among the randomly amplified polymorphic DNA method, multilocus enzyme electrophoresis, and southern blot hybridization with the moderately repetitive DNA probe Ca3 for fingerprinting Candida albicans. J Clin Microbiol 1997; 35(9): 2348-58. [Özet] [PDF]
- Valério HM, Weikert-Oliveira Rde C, Resende MA. Differentiation of Candida species obtained from nosocomial candidemia using RAPD-PCR technique. Rev Soc Bras Med Trop 2006; 39(2): 174-8. [Özet]
- McCullough MJ, Clemons KV, Stevens DA. Molecular and phenotypic characterization of genotypic Candida albicans subgroups and comparison with Candida dubliniensis and Candida stellatoidea. J Clin Microbiol 1999; 37(2): 417-21. [Özet] [Tam Metin] [PDF]
-
Soll DR. The ins and outs of DNA fingerprinting the infectious fungi.
Clin Microbiol Rev 2000; 13(2): 332-70.
[Özet] [Tam Metin] [PDF] - Karahan ZC, Akar N. Subtypes of genotype A Candida albicans isolates determined by restriction endonuclease and sequence analyses. Microbiol Res 2005; 160(4): 361-6. [Özet]
- Kaiser C, Michaelis S, Mitchell A. Methods in Yeast Genetics, pp: 137-47. 1994. Cold Spring Harbor Laboratory Press, New York.
- Hunter PR, Gaston MA. Numerical index of the discriminatory ability of typing systems: an application of Simpson's index of diversity. J Clin Microbiol 1988; 26(11): 2465-6. [Özet] [PDF]
- Hattori H, Iwata T, Nakagawa Y, et al. Genotype analysis of Candida albicans isolates obtained from different body locations of patients with superficial candidiasis using PCRs targeting 25S rDNA and ALT repeat sequences of the RPS. J Dermatol Sci 2006; 42(1): 31-46. [Özet]
- Qi QG, Hu T, Zhou XD. Frequency, species and molecular characterization of oral Candida in hosts of different age in China. J Oral Pathol Med 2005; 34(6): 352-6. [Özet]
- Iwata T, Hattori H, Chibana H, et al. Genotyping of Candida albicans on the basis of polymorphisms of ALT repeats in the repetitive sequence (RPS). J Dermatol Sci 2006; 41(1): 43-54. [Özet]
-
Garcia-Hermoso D, Cabaret O, Lecellier G, et al. Comparison of
microsatellite length polymorphism and multilocus sequence typing for DNA-based
typing of Candida albicans. J Clin Microbiol 2007; 45(12): 3958-63.
[Özet] [Tam Metin] [PDF] -
Robles JC, Koreen L, Park S, Perlin DS. Multilocus sequence typing is a
reliable alternative method to DNA fingerprinting for discriminating among
strains of Candida albicans. J Clin Microbiol 2004; 42(6): 2480-8.
[Özet] [Tam Metin] [PDF] - Dassanayake RS, Ellepola ANB, Samaranayake YH, Samaranayak LP. Molecular heterogeneity of fluconazole-resistant and -susceptible oral Candida albicans isolates within a single geographic locale. APMIS 2002; 110(4): 315-24. [Özet]
- Metzgar D, van Belkum A, Field D, Haubrich R, Wills C. Random amplification of polymorphic DNA and microsatellite genotyping of pre- and posttreatment isolates of Candida spp. from human immunodeficiency virus-infected patients on different fluconazole regimens. J Clin Microbiol 1998; 36(8): 2308-13. [Özet] [Tam Metin] [PDF]
- Wilson MJ, Williams DW, Forbes MD, Finlay IG, Lewis MA. A molecular epidemiological study of sequential oral isolates of Candida albicans from terminally ill patients. J Oral Pathol Med 2001; 30(4): 206-12. [Özet]
İletişim (Correspondence):
Doç. Dr. Zeynep Ceren Karahan,
Ankara Üniversitesi Tıp Fakültesi,
Tıbbi Mikrobiyoloji Anabilim Dalı,
Morfoloji Binası,
Sıhhiye, 06100, Ankara, Türkiye.
Tel (Phone): +90 312 595 8120,
E-posta (E-mail): ckarahan@medicine.ankara.edu.tr