Salmonella Serotip Enteritidis
İzolatlarının Plazmid Profil Analizi ve Pulsed Field
Jel Elektroforezi ile İncelenmesi
Investigation
of Salmonella Serotype Enteritidis Isolates By Plasmid Profile Analysis
and
Pulsed Field Gel Electrophoresis
Ebru
US, Birsel ERDEM, Alper TEKELİ, Devran GERÇEKER, Begüm SARAN, Mehseti
BAYRAMOVA,
Fikret ŞAHİN
Ankara Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Ankara.
Ankara University Faculty of Medicine, Department of Medical Microbiology, Ankara, Turkey.
ÖZET
Bu çalışmada, Salmonella serotip Enteritidisin moleküler epidemiyolojisinin aydınlatılması amacıyla, Ankara Üniversitesi Tıp Fakültesi Tıbbi Mikrobiyoloji Anabilim Dalı, Enterobakteri Laboratuvarının kültür koleksiyonlarından seçilen 122 Salmonella Enteritidis suşu, Kado ve Liu yöntemine göre plazmid profil analizi (PPA) ile ve Dünya Sağlık Örgütü protokollerine göre, SpeI ve XbaI enzimleri kullanılarak pulsed field jel elektroforezi (PFGE) ile incelenmiştir. Çalışmaya dahil edilen suşlar, 2000 yılından sonra 10 farklı ildeki klinik mikrobiyoloji laboratuvarında, salgın (n= 13) ve sporadik (n= 109) olgulara ait klinik örneklerden (103 dışkı, 16 kan ve birer adet safra, idrar ve beyin omurilik sıvısı) izole edilen suşlardır. PFGE ile elde edilen bant sayı ve ilişkileri GeneDirectory (Syngene, İngiltere) jel analiz programı kullanılarak; küme analizi Dice benzerlik katsayısı ve UPGMA (Unweighted Pair Group Method Average) ilişki kuralı parametreleriyle değerlendirilmiştir. Çalışmada, plazmid taşıyan 110 (%90) suşun, büyüklükleri 2.0 ile 100 kb arasında değişen 1-4 plazmid taşımakta olduğu ve 14ten fazla plazmid profili (p1-p14) gösterdiği belirlenmiştir. Seksen beş (%69.7) izolatın, 57 kb büyüklüğündeki plazmidi tek olarak ya da başka plazmidlerle birlikte bulundurduğu izlenmiştir. PFGE yönteminde SpeI ve XbaI enzimleri ile 11er farklı PFGE modeli elde edilmiş; SpeI ile oluşan modeller A-K; XbaI ile oluşanlar a-k şeklinde adlandırılmıştır. SpeI ile model Ada 93 (%76.2) suşun; XbaI ile model ada 53 (%43.4) ve model bde 42 (%34.4) suşun kümelendiği belirlenmiştir. İki enzim birlikte değerlendirildiğinde ortaya çıkan 17 farklı PFGE kümesi içinde, suşların çoğunluğunun Aa (50 suş, %40.9) ve Ab (42 suş, %34.4) kümelerinde toplandığı görülmüştür. PFGE yönteminde SpeI ve XbaI enzimlerinin birlikte kullanılması ile ayırım gücünün arttığı belirlenmiş ve bulgularımız Türkiyede dolaşımdaki S.Enteritidis izolatlarının çoğunluğunun benzer PFGE modelleri gösteren suşlar olduğunu ortaya çıkarmıştır. Bu nedenle S.Enteritidis suşlarının PFGE ile araştırılması durumunda, en azından plazmid profil analizi gibi bir başka yöntemle beraber uygulanması, izolatların ilişkilerini ortaya çıkarmak için daha yararlı olacaktır.
Anahtar sözcükler: Salmonella serotip Enteritidis; plazmid profil analizi; PFGE.
ABSTRACT
In this study a total of 122 Salmonella serotype Enteritidis stock strains selected from the culture collection of Enterobacteriaceae Laboratory of Ankara University Faculty of Medicine, Department of Medical Microbiology, were investigated by plasmid profile analysis with the method defined by Kado and Liu and pulsed field gel electrophoresis (PFGE) according to World Health Organization protocols using SpeI and XbaI macrorestriction enzymes, for better understanding of the molecular epidemiology of S.Enteritidis. The study strains were selected from a collection of previously isolated epidemic (n= 13) and sporadic (n= 109) strains (103 stool, 16 blood and one each bile, urine and cerebrospinal fluid) obtained from 10 different cities after the year 2000. PFGE patterns were analyzed with Gene Directory software (Syngene, UK) and a similarity index was determined by using Dice coefficient and the unweighted pair group method with mathematical averaging (UPGMA). Plasmid-carrying 110 (90%) strains that harbored 1-4 plasmids with sizes ranging from 2.0 to 100 kb were separated into patterns more than 14 (p1-p14). A total of 85 (69.7%) isolates harbored the 57 kb plasmid solely or in combination with other plasmids. By PFGE, 11 distinct patterns were shown with each enzyme SpeI and XbaI. S. Enteritidis strains after digestion with macrorestriction enzyme SpeI generated 11 different PFGE patterns (A to K), whereas XbaI generated also 11 different PFGE patterns (a to k). PFGE pattern A consisted of 93 strains (76.2%) after digestion with macrorestriction enzyme SpeI, while PFGE pattern a consisted 53 (43.4%) and PFGE pattern b 42 strains (34.4%) after digestion with macrorestriction enzyme XbaI. Using two macrorestriction enzymes two PFGE cluster profiles Aa (50 strains, 40.9%) and Ab (42 strains, 34.4%) were found to be predominating among 17 different PFGE clusters. Our results confirmed the clonal nature of S.Enteritidis strains in Turkey. The use of two enzymes in PFGE analysis appeared to increase the discriminatory power of PFGE, leading to greater diversity among strains. PFGE analysis performed by SpeI and XbaI enzymes combined with plasmid profiling could be established as a useful tool for detection of genetic relationship between isolates.
Key words: Salmonella serotype Enteritidis; plasmid profile analysis; PFGE.
Geliş Tarihi (Received): 18.10.2010 • Kabul Ediliş Tarihi (Accepted): 14.02.2011
GİRİŞ
Salmonella enfeksiyonları Türkiyede önemli bir halk sağlığı sorunudur1. Dünyada son 25 yılda Salmonella serotiplerinden Salmonella enterica subsp. enterica ser. Enteritidis belirgin bir şekilde yaygınlaşırken, Türkiyede de 1990lı yıllardan beri insan enfeksiyonlarında en sık karşılaşılan serotip olmuştur2,3.
Salmonella serotip Enteritidis gastroenteritli olguların örneklerinden sıklıkla izole edilen serotip olduğu gibi, bağırsak dışı invazif enfeksiyonlara da yol açabilen önemli bir etkendir. Gıda kaynaklı salgınlara neden olduğu kadar sporadik olgularda da en sık rastlanan Salmonella serotipidir3. S.Enteritidisin, Batı Avrupada Enterik Enfeksiyonların Ulusal Sürveyans Ağı (Enter-net)na ait ulusal referans laboratuvarlarında en sık tanımlanan serotip olduğu bildirilmektedir4,5.
Salmonella cinsindeki bakteriler, lipopolisakkarid O (somatik) antijenleri ve protein H (kirpik) antijenlerinin farklılıklarına dayanılarak, 1926 yılında Whiteın düzenlediği ve 1972-1978 yıllarında Kauffmannın genişlettiği şemaya göre serogruplara ve serotiplere ayrılır1. Salmonella izolatları, Türkiyede çoğu çalışmada serotiplendirme ve antimikrobiyal duyarlılık testleriyle araştırılmıştır6. İzolatların çoğunun benzer antibiyogramlar gösterdiği epidemiyolojik çalışmalarda, bu fenotipik yöntem Salmonella serotiplerini tiplendirmede yeterli ayırt edici güce sahip değildir. Epidemiyolojik çalışmalarda Salmonella serotiplerinin alttiplendirmesi (subtyping) için ve izolatların aralarındaki ilişkinin gösterilebilmesi amacıyla standart tekniklere ihtiyaç vardır. Son yıllarda Salmonella izolatlarının ileri ayırımı için moleküler tiplendirme yöntemleri geliştirilmiştir7. Plazmid ve kromozom DNAsının incelenmesine dayanan plazmid analizi, ribotiplendirme, pulsed field jel elektroforezi (PFGE) gibi moleküler yöntemler her geçen gün geliştirilmekte ve yeni yöntemler eklenmektedir8. PFGE, Salmonella suşlarının ayırımında kullanılan moleküler yöntemler içinde yaygın olarak önerilen ve Amerika Birleşik Devletlerinde 1996 yılından beri Salmonella alttiplendirmesinde uygulanan başlıca yöntemdir9.
Bu çalışmada, S.Enteritidisin moleküler epidemiyolojisine katkıda bulunmak üzere Türkiyede salgın ve sporadik olgulardan izole edilen S.Enteritidis suşlarının plazmid profil analizi (PPA) ve iki enzim (SpeI ve XbaI) kullanılarak PFGE yöntemleriyle araştırılması amaçlanmıştır.
GEREÇ ve YÖNTEM
Suşlar
Ankara Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı Enterobakteri Laboratuvarının kültür koleksiyonlarında bulunan 122 S.Enteritidis suşu çalışmaya alındı. Bu suşlar, daha önceki bir çalışma kapsamında3 2000 yılından itibaren, 10 ildeki [Ankara (n= 43), Antalya (n= 2), Bursa (n= 12), Edirne (n= 27), Eskişehir (n= 7), İstanbul (n= 2), İzmir (n= 2), Kayseri (n= 17), Konya (n= 8), Trabzon (n= 2)] klinik mikrobiyoloji laboratuvarlarında, salgın (n= 13) ve sporadik (n= 109) olgulara ait klinik örneklerden [dışkı (n= 103), kan (n= 16), safra (n= 1), idrar (n= 1), beyin omurilik sıvısı (BOS) (n= 1)] standart yöntemlerle izole edilmiş; Ankara Üniversitesi Tıp Fakültesi Tıbbi Mikrobiyoloji Anabilim Dalında serotiplendirilmiş; Clinical and Laboratory Standards Institute (CLSI; eski adı ile NCCLS) kılavuzunda açıklandığı şekilde agar dilüsyon yöntemiyle antibiyotik duyarlılıkları belirlendikten sonra, mikrobank kültür saklama tüpleri içinde -80°Cde saklanmakta olan izolatlardı. Bu suşlar, yaşları 1-90 yıl arasında değişen 101 gastroenterit, 13 sepsis, 8 paratifo olgusundan (38 kadın, 84 erkek) izole edilmişti. Bu izolatların 13ü, Edirne yakınlarındaki bir askeri birlikte meydana gelen gıda kaynaklı bir salgın sırasında 20-22 yaşlarındaki gastroenteritli olguların dışkı kültürlerinden elde edilen suşlardı.
Plazmid Eldesi ve Plazmid Profil Analizi
Plazmidler, Kado ve Liunun10 yönteminde küçük değişiklikler yapılarak elde edildikten sonra, 0.5 X Tris-borik asit-EDTA (TBE) solüsyonundaki 0.5 µg etidyum bromür ml-1 içeren %0.7lik horizontal agaroz jelde (Serva, Heidelberg, Almanya) 100 Vda iki saat yürütülerek ayrıldı. İncelenen suşların plazmid büyüklükleri, plazmid büyüklükleri bilinen Escherichia coli V517 suşu (53.7, 7.2, 5.6, 5.1, 3.9, 3.0, 2.7, 2.1 kb), Salmonella Typhimurium 020255-Ankara suşu (90 kb), Salmonella Enteritidis 006956-Ankara (57, 5.8, 4.8 kb) suşu ve bir moleküler büyüklük belirteci (gene ruler 1 kb DNA Ladder, Fermentas, Vilnius, Litvanya) ile birlikte elektroforez uygulanarak belirlendi. Plazmid DNA bantları, UV ışığı altında (TFX 20M-Vilber Lourmat, Marna la Vallee, Fransa) görüntülendi. Kontrol suşları (E.coli V517, S. Typhimurium 020255-Ankara ve S.Enteritidis 006956-Ankara) Refik Saydam Hıfzıssıhha Merkezi Bulaşıcı Hastalıklar Araştırma Bölümünden sağlandı. İzolatların plazmid DNA içerikleri kilobaz (kb) şeklinde molekül ağırlıklarına uygun olarak tanımlandı.
PFGE
PFGE, Dünya Sağlık Örgütü (DSÖ) protokolüne göre uygulandı11. Bu yönteme göre her bir izolat SpeI ve XbaI enzimleri (Fermentas, Vilnius Litvanya) ile ayrı ayrı kesim işlemine tabii tutuldu. Tüm izolatlar bir gece 37°Cde üretildikten sonra %1.3lük düşük erime ısılı agaroz ile agaroz kalıpları hazırlandı. Genomik DNA, 20 Ulik kesim enzimleri SpeI ve XbaI ile üreticinin önerileri doğrultusunda dört saat inkübe edildi. CHEF DR II (Bio-Rad, Hercules, CA, USA) sisteminde, %1.3lük agaroz jel ile uygulanan elektroforez koşulları başlangıç vuruş süresi 2.2 saniye, bitiş vuruş süresi 63.8 saniye, akım 6 V/cm, sıcaklık 14°C, süre 18 saat olacak şekilde ayarlandı. Jeller etidyum bromür (0.2 µg/ml) solüsyonunda boyandı ve UV ışığı altında görüntülendi. Elektroforez sonrasında jel fotoğrafları DigiGenius görüntüleme sistemi (Syngene-UK) kullanılarak çekildi. PFGE paternleri, hem Tenover ve arkadaşları12 tarafından tarif edildiği şekilde gözle, hem de Gene Directory (Syngene, Cambridge, İngiltere) programı kullanılarak değerlendirildi. Unweighted Pair Group Method with Mathematical Averaging (UPGMA) yöntemi ve Dice benzerlik katsayısı kullanılarak PFGE paternlerinin %1lik bant toleransı ile dendrogramı oluşturuldu ve kümelenme analizi yapıldı. Kontrol olarak bir moleküler büyüklük belirteci (M) [N0340S, New England Biolabs, Hetfordshire, İngiltere (Band büyüklüğü 48.5-1018.5 kb)] kullanıldı. Kontrol suşu (Salmonella Braenderup H9812) Refik Saydam Hıfzıssıhha Merkezi Başkanlığı Bulaşıcı Hastalıklar Araştırma Bölümünden sağlandı.
BULGULAR
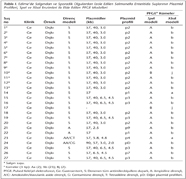
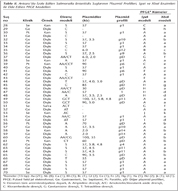
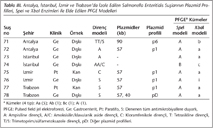
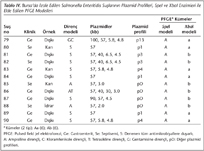
İzolatların antimikrobiyal duyarlılıkları, plazmid profilleri, PFGE modelleri (paternleri) ve diğer özellikleri Tablo I, II, III, IV, V, VI, VIIde gösterilmiştir.
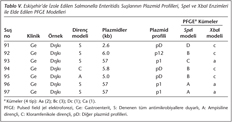
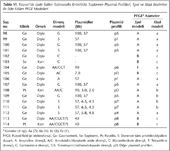
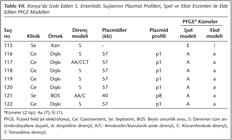
İncelenen 122 S.Enteritidis suşunun 12 (%10)sinin plazmidsiz suşlar olduğu görülmüştür. Plazmid taşıyan 110 (%90) izolatın, büyüklükleri 2.0-100 kb arasında değişen 1-4 plazmid taşıdığı belirlenmiştir (Tablo I, II, III, IV, V, VI, VII). Seksen beş (%69.7) izolat 57 kb büyüklüğündeki plazmidi tek olarak ya da başka plazmidlerle birlikte taşımaktadır (Tablo I, II, III, IV, V, VI, VII, VIII, IX). Plazmidli suşların 14ten fazla plazmid profili (p1-p14) gösterdiği belirlenmiştir (Tablo VIII). Plazmid profillerinin çoğunda (p1-p5, p7, p9-p11, p13) 57 kblık plazmid bulunmaktadır ve izolatların çoğu (n= 34, %27.9) p1 profiline sahiptir (Tablo VIII). Ankarada S.Enteritidis suşlarından elde edilen altı plazmid (85, 70, 60, 55, 4.0, 3.5 kb), diğer illerin izolatlarında görülmemiştir (Tablo II ve Tablo IX). Sadece Edirnede belirlenen p2 profili (57, 40, 3.0 kb) gösteren suşların 13ü gıda kaynaklı salgın suşlarıdır (Tablo I). Ayrıca, p5 profili (100, 57 kb) sadece Kayseri suşlarında belirlenmiştir (Tablo VI).
S.Enteritidis suşlarının PFGE yöntemi ile SpeI ve XbaI makrorestriksiyon enzimleri ile kesimlerinin ardından, molekül büyüklükleri her iki enzimle de 48.5-1018.5 kb arasında değişen 8-13 adet bant elde edilmiştir. Dice benzerlik katsayısı ve UPGMA yöntemi kullanılarak oluşturulan PFGE modellerinin dendogramları sırasıyla Şekil 1 ve 3te gösterilmiştir.
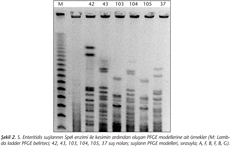
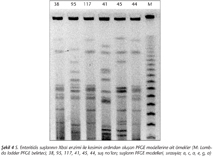
SpeI enzimi ile kesilen S.Enteritidis suşları 8-13 bant içeren 11 farklı PFGE modeli (A-K) oluşturmuştur. İki ana PFGE modeli olan A ve Bnin genetik benzerliklerinin %38 olduğu gösterilmiştir. İzolatların 93 (%76.2)ü PFGE modeli Ayı, 12 (%9.8)si PFGE model Byi oluşturmuştur (Şekil 1, Tablo X). Kalan 17 izolatın oluşturduğu dokuz PFGE modeli (C-K) üçten fazla bant farkı ve %80in altında benzerlik göstermiştir. S.Enteritidis suşlarının SpeI enzimi ile kesiminin ardından elde edilen PFGE modellerine ait örnekler Şekil 2de verilmiştir.
XbaI enzimi ile kesilen S. Enteritidis suşları 8-13 bant içeren 11 farklı PFGE modeli (a-k) oluşturmuştur. Üç ana PFGE modeli a, b ve c, sırasıyla tüm izolatların %43.4, %34.4 ve %9unda saptanmıştır (Şekil 1, Tablo X). Kalan sekiz PFGE modeli 16 izolattan elde edilmiş; bu modeller arasında üçten fazla bant farkı ve %80in altında benzerlik gözlenmiştir. PFGE model a ve model b arasındaki genetik benzerlik %91den fazladır. Bu iki PFGE modeli ile model c arasındaki genetik benzerlik ise %10dur (Şekil 3). S.Enteritidis suşlarının XbaI enzimi ile kesiminin ardından elde edilen PFGE modellerine ait örnekler Şekil 4te gösterilmiştir.
İki enzim birlikte değerlendirildiğinde, S.Enteritidis suşlarının 17 farklı PFGE kümesine ayrıldığı görülmüştür (Tablo X). Üç ana küme olan Aa, Ab, Bcnin tüm izolatlar arasındaki dağılımı sırasıyla %40.9, %34.4 ve %8.1 olarak belirlenmiştir.
TARTIŞMA
Türkiyede en sık izole edilen Salmonella enterica serotipi S.Enteritidisdir1,3. Dünyanın pek çok bölgesinde de yaygın olduğundan S.Enteritidis enfeksiyonları küresel bir pandemi olarak kabul edilmektedir13. 1997 yılında Avrupada bildirilen tüm insan salmonelloz olgularının %85i S. Enteritidis kaynaklı iken, bu yüksek seviyeden itibaren insidansın düştüğü gözlenmiştir7. Avrupada gıda kaynaklı enfeksiyonların en sık nedeni Salmonellalardır ve S.Enteritidis de en sık tanımlanan serotiptir2. İngiltere ve İspanyadan yapılan son çalışmalar S.Enteritidisin sırasıyla %60 ve %86 insidans ile en sık rastlanan serotip olduğunu göstermiştir. 2000-2002 yılları arasında S.Enteritidis Avrupada Enter-nete bildirilen suşların %53ünü oluşturmuştur14.
Avrupada salgınların incelemesinde, Salmonella serotiplerinde suşların ileri ayırımı (alttiplendirme) gerektiğinde, DNA parmak izi incelemesiyle beraber yapılan Ward ve arkadaşlarının15 yöntemine dayanan faj tiplendirme, uluslararası standart bir yöntem olarak kullanılmaktadır2. Geniş konak çeşitliliğine sahip bir patojen olan S.Enteritidis faj tipleri, çeşitli bölgelerde ve hayvan türlerinde bildirilmiştir13. Erdem ve arkadaşları16 1994 yılında Türkiyede faj tip 4e (PT4) ait suşların hakimiyetini göstermişlerdir. Daha sonra Avrupada 1998-2003 yılları arasında insan izolatlarına ait faj tiplerinde değişiklik olduğu, PT4te azalma ve PT8 ile PT21in yaygınlığında artış olduğu bildirilmiştir5. Plazmid ve kromozom DNAsının incelenmesine dayanan yöntemler, faj tiplendirmeye ek olarak kullanılmaktadır16,17,18. Geleneksel faj tiplendirme yöntemi zaman alıcıdır, faj süspansiyonlarının devam ettirilmesi güçtür ve belirli referans merkezlerinde uygulanabilmektedir13.
Günümüzde etkenin belli bölgelerde endemik olması nedeniyle, epidemiyolojik sürveyans için duyarlı ve standart yöntemler gereklidir19. Moleküler tiplendirme, Salmonella sürveyansının duyarlılık ve özgüllüğünü büyük ölçüde artırmakta8,20 ve geleneksel sürveyans yöntemleriyle saptanamayacak salgınları belirleme ve halk sağlığı için önemli özel klonları hızla tanımlama olanağı sağlamaktadır13. PPA (plasmid profile analysis), RAPD (randomly amplified polymorphic DNA), IS200 RFLP (restriction fragment length polymorphism), ribotiplendirme ve PFGE muhtemel genetik ilişkileri belirleme ve tanımlamada kullanılan yöntemlerdir21.
Plazmidler, çoğunlukla kovalan olarak kapalı, dairesel yapıda, çift iplikçikli kromozom DNA molekülleridir ve bakteri kromozomundan bağımsız olarak replike olur. Epidemiyolojik amaçlar için uygulanan PPA, suşlardaki plazmid DNAsının ekstraksiyonundan sonra her bir suşun taşıdığı plazmidlerin sayı ve moleküler ağırlıklarının belirlenmesi temeline dayanır. Çeşitli Salmonella salgınlarının aydınlatılmasını sağlamasına karşın PPA, plazmid taşıyan suşlarla sınırlıdır. S.Enteritidis izolatlarının çoğu çevresel konaklarda devam edebilmesi için önemli olduğu düşünülen 38 MDaluk serovara özgül virülans plazmidini taşımaktadır. Salmonella serotiplerinin alttiplendirmesinde, suşların plazmid taşımadığı; suşların sadece serotipe özgü plazmid taşıdığı ya da suşların çok azının plazmidi olduğu durumlarda PPA faydalı olamamaktadır16,22.
Çalışmamızda PPA, incelenen 122 S.Enteritidis izolatının 110 (%90)unun plazmidli izolatlar olduğunu göstermiştir. S. Enteritidis suşlarının çoğunun taşıdığı bilinen 38 MDaluk serovara özgül virülans plazmidine karşılık gelen büyüklükteki 57 kblık plazmidin, çalışmamızda 34 (%27.9) izolatta tek başına (plazmid profili p1) bulunduğu gösterilmiştir (Tablo VIII). Buna karşın incelenen izolatların 14ten fazla plazmid profiline dağıldıkları belirlenmiştir. Bununla birlikte PFGE modellerinin her zaman tek bir plazmid profiline karşılık gelmediği görülmüştür. Plazmid profillerinden p1 (57 kb) ve p4 (57, 5.8 ve 4.8 kb) ile PFGE kümesi Aa arasında; Edirnede görülen plazmid profili p2 (57, 40, 3.0 kb) ile PFGE kümesi Ab arasında; ayrıca plazmid profili p3 (57, 40, 6.5, 4.5 kb) ile PFGE kümesi Ab arasında güçlü bir ilişki bulunduğu düşünülmektedir (Tablo I, II, III, IV, V, VI, VII).
PFGE, ilk tarif edildiği 1983 yılından sonra birçok bakteri için kullanılan moleküler epidemiyolojik yöntemler içinde, suşlar arasındaki genetik ilişkiyi ortaya koymada güvenilir standart bir yöntem haline gelmiştir. PFGE, günümüzde Salmonella serotipleri için de standart bir yöntem olarak kabul edilmektedir14. Bu teknikte, megabaz büyüklüğündeki kromozom DNAsı, seyrek olarak, aralıklı kesen makrorestriksiyon enzimleriyle kesilmekte; kesilen büyük DNA parçaları, farklı yönlerden elektrik akımı uygulayabilen elektroforez aygıtı ile jel üzerinde yürütüldükten sonra görüntülenebilmektedir12. PFGE, ABDde ve çeşitli Avrupa ülkelerinde Salmonella salgın araştırmalarında elde edilen bulguları karşılaştırabilmek amacıyla standart protokolle uygulanmaktadır8,14. PFGEnin XbaI ile kullanımı, Salmonella serotipleri için yaygın olarak kabul görmektedir23,24. Bazı çalışmalarda, kontamine etten insana Salmonella bulaşının gösterilmesi örneğinde olduğu gibi enfeksiyon zinciri PFGE ile doğrulanmıştır7,16.
2001 yılında kurulan uluslararası bir proje olan Avrupa Birliği DG Araştırma-Fonu girişimi Salm-gene tarafından, PFGE modellerinin (paternlerinin) gerçek zamanlı olarak onaylanması ve ileri sürveyans için kriterlerin belirlenmesi amacıyla, dokuz Avrupa ülkesindeki insan izolatlarından elde edilen PFGE modellerine ait taranabilir bir veri bankası geliştirilmiştir. En sık rastlanan PFGE modelleri olan SENTXB.0001 (%56), SENTXB.0002 (%16) ve SENTXB.0005 (9.5%) veri bankasına gönderilen tüm S. Enteritidis izolatlarının neredeyse %82sini oluşturmaktadır. Peters ve arkadaşlarına2 göre, PFGE modellerinin yüksek derecede benzerliği ve dağılımının ortaya koyduğu bu veriler, S.Enteritidisin klonal yapısını vurgulamaktadır2. Bu bulgu, tüm hayvan ve insan izolatlarının %64ünün tek bir PFGE modelinde toplandığının belirlendiği diğer bir çalışma ile uyumlu bulunmaktadır25. Çalışmamızda da, Salm-gene projesinde görüldüğü gibi S.Enteritidis izolatlarının belirli PFGE kümelerinde (Aa ve Ab) toplanması (Tablo X), bu izolatlarının klonal olduğunu düşündüren verilerle uyumludur.
Çeşitli çalışmalar, PFGEde BlnI gibi ikinci bir enzim veya birden fazla teknik kullanıldığında S.Enteritidis izolatlarının daha fazla ayırt edilebildiğini göstermiştir2,26. PFGEde ikinci bir kesim enziminin kullanılması, belli serotiplerde ve S.Enteritidisin PT4 gibi hakim faj tiplerinin ayırt edilmesinde faydalı görülmektedir2. Çalışmamızda da, S.Enteritidis klinik izolatlarının tiplendirilmesinde iki kesim enziminin kullanılmasının, tek bir enzimin kullanılmasına göre biraz daha ayırt edici olduğu görülmüştür. Çalışmamızda SpeI ve XbaI enzimlerinin her biri ile 11 PFGE modeli; her iki enzim birlikte değerlendirildiğinde ise 17 farklı PFGE kümesi elde edilmiştir. SpeI enzimi ile 11 farklı model arasında bir model (A) esas olarak hakimken (93 izolat; %76.2); XbaI enzimi ile elde edilen bantların dendogramında %91den fazla genetik benzerliğe (muhtemel ilişkili) sahip olduğu belirlenen iki modelin (a ve b) hakim olduğu görülmüştür (sırasıyla; 53 izolat, %43.4 ve 42 izolat, %34.4). Her iki enzim birlikte değerlendirildiğinde ise 17 farklı PFGE kümesi içinde iki kümenin (Aa ve Ab) hakim olduğu ortaya çıkmıştır (sırasıyla; 50 izolat, %40.9 ve 42 izolat, %34.4) (Tablo X).
Çalışmamızda, 2000 yılında Edirne yakınlarındaki askeri birlikte meydana gelen gıda kaynaklı salgından elde edilen, antibiyotiklere duyarlı 13 izolatın tümünün benzer plazmid profilini (p2) gösteren ve biri dışında benzer PFGE kümesine (Ab) ait olan izolatlar olduğu görülmüştür. Çalışmada elde edilen bu moleküler bulgular, PFGE kümesi Bjye ait suşun (suş no. 10) gerçek bir salgın izolatı olmadığını, izole edildiği olgunun aynı yerde, aynı zaman diliminde benzer semptomları göstermesinden dolayı izolatın salgın şüpheli suşlara dahil edildiğini göstermiştir. Bu bulgular, S.Enteritidise ait salgın suşlarının ayırt edilmesinde, PPA ve PFGEnin yararlı olduğunu göstermiştir (Tablo I).
Türkiyede izole edilen suşların incelendiği bir çalışmada, 2001-2004 yılları arasında toplanan ve genetik olarak benzer olmayan 26 S.Enteritidis suşunun %92sinin aynı plazmidi (38 MDa) taşıdığı görülürken, XbaI enzimi ile kesimin ardından tip I, II, III ve IV olmak üzere dört PFGE kümesi elde edilmiştir21. Rivoal ve arkadaşlarının7 çalışmasında, üç örnekleme periyodu boyunca üç farklı yumurta fabrikasından toplanan 144 sıvı yumurta örneğinden elde edilen S. Enteritidis izolatları, genomik DNAnın XbaI ve SpeI enzimleri ile kesimini takiben PFGE ile tiplendirilmiş ve S. Enteritidisin klonal yapıda olduğunu doğrulayacak şekilde benzerlik oranları yüksek olan (%88) üç küme belirlenmiştir. Bu durum pek çok çalışmada da gösterilmiştir27,28,29,30,31. Pang ve arkadaşları32,33 Tayvan ve Almanyada S.Enteritidis insan izolatlarına ait genomların 13 yıl boyunca (1991-2002) çok iyi korunduğu sonucuna varmışlardır. Murase ve arkadaşları34 ise, Salmonella serotiplerinin PFGE alt tiplendirmesinde iki makrorestriksiyon enzimiyle yapılan incelemenin daha ayırt edici olduğunu göstermişlerdir. Çalışmamızda da, iki enzimi (SpeI ve XbaI) bir arada değerlendirdiğimizde daha fazla sayıda PFGE kümeleri ortaya çıkmıştır. Ayrıca benzer direnç modeli gösteren S.Enteritidis suşlarının, PPA ve iki enzimli PFGE ile kolayca ayrılabildiği görülmüştür (Tablo I, II, III, IV, V, VI, VII). Yapılan çalışmalarda, farklı kaynaklardan elde edilen S.Enteritidis izolatlarının alttiplendirilmesinde PPA ve PFGEnin faydalı yöntemler olduğu rapor edilmektedir13,33. Çalışmamızda, sırasıyla tüm izolatların %40.9, %34.4 ve %8.1ini oluşturan Aa, Ab ve Bc kümelerindeki S.Enteritidis izolatları, antibiyotik direnç modelleri ve plazmid profilleri açısından farklı bulunmuştur (Tablo I, II, III, IV, V, VI, VII). Direnç modelleri ve genotipler arasında ilişki saptanmazken, antibiyotiklere çoklu direnç gösteren izolatların farklı PFGE kümelerine ve farklı plazmid profillerine dağıldığı belirlenmiştir.
Sunulan bu çalışma, SpeI ve XbaI enzimleri ile uygulanan PFGEnin, PPA ile birlikte değerlendirildiğinde, benzer direnç fenotipine sahip izolatların ayırt edilmesinde ve farklı direnç modellerine sahip izolatlar arasındaki genetik ilişkiyi saptamada yararlı olduğunu; iki enzimle elde edilen modellerin birlikte değerlendirilmesiyle, PFGE yönteminin S.Enteritidis için ayırt ediciliğinin arttığını göstermektedir. Ayrıca bulgularımız, Türkiyede dolaşımdaki S.Enteritidis izolatlarının çoğunluğunun benzer PFGE modelleri gösteren suşlar olduğunu ortaya çıkarmıştır. Bu bulgular, S.Enteritidis suşlarının klonal yapısını doğrular niteliktedir. S.Enteritidis izolatlarının incelenmesinde SpeI ve XbaI enzimleri ile yapılan PFGE, plazmid profilleri ile beraber ele alındığında ayırt ediciliği yüksek, güvenilir bir yöntem olarak değerlendirilmekle birlikte, S.Enteritidis suşlarının moleküler epidemiyolojisi ile ilgili bilgilerin güncellenmesi için sürekli yeni çalışmaların yapılması gereklidir.
TEŞEKKÜR
Çalışmada izolatları sağlayan öğretim üyelerine [A.D. Aysev, Ankara Üniversitesi Tıp Fakültesi (ÜTF); G. Hasçelik, D. Gür ve S. Ercis, Hacettepe ÜTF; S. Gedikoğlu, Uludağ ÜTF; B. Sümerkan ve D. Eşel, Erciyes ÜTF; İ. Tuncer, Selçuk ÜTF; M. Tuğrul ve M. Tatman-Otkun, Trakya ÜTF; A. Tünger, Ege ÜTF; Y. Akgün, Osmangazi ÜTF; M. Gültekin, Akdeniz ÜTF; İ. Köksal, Karadeniz Teknik ÜTF; G. Söyletir, Marmara ÜTF] ve kontrol suşlarını [Salmonella Braenderup (H9812), E.coli (V517), Salmonella Typhimurium (020255-Ankara) ve Salmonella Enteritidis (006956-Ankara)] sağlayan Refik Saydam Hıfzıssıhha Merkezi Başkanlığı Bulaşıcı Hastalıklar Araştırma Bölümünden B. Esen ve B. Levente teşekkür ederiz.
KAYNAKLAR
- Toreci K, Ang O. Türkiyede saptanmış olan Salmonella serovarları ve salmonellozların genel değerlendirilmesi. Türk Mikrobiyol Cem Derg 1991; 21(1): 1-18.
- Peters TM, Berghold C, Brown D, et al. Relationship of pulsed-field profiles with key phage types of Salmonella enterica serotype Enteritidis in Europe: results of an international multi-centre study. Epidemiol Infect 2007; 135(8): 1274-81. [Özet] [Tam Metin] [PDF]
- Erdem B, Hasçelik G, Gedikoğlu S, et al. Salmonella enterica serotipleri ve Salmonella enfeksiyonları: Türkiyede on ili kapsayan çok merkezli bir çalışma. Mikrobiyol Bul 2004; 38(3): 173-86. [Özet] [PDF]
- Fisher IS. The Enter-net international surveillance network-how it works. Euro Surveill 1999; 4(5): 52-5. [Özet] [Tam Metin]
- Fisher IS and Enter-net participants. Dramatic shift in the epidemiology of Salmonella enterica serotype Enteritidis phage types in western Europe, 1998-2003-results from the Enter-net international salmonella database. Euro Surveill 2004; 9(11): 43-5. [Özet] [Tam Metin]
- Aysev AD, Güriz H, Erdem B. Drug resistance of Salmonella strains isolated from community infections in Ankara, Turkey, 1993-99. Scand J Infect Dis 2001; 33(6): 420-2. [Özet]
- Rivoal K, Protais J, Quéguiner S, et al. Use of pulsed-field gel electrophoresis to characterize the heterogeneity and clonality of Salmonella serotype Enteritidis, Typhimurium and Infantis isolates obtained from whole liquid eggs. Int J Food Microbiol 2009; 129(2): 180-6. [Özet]
- Liebana E, Garcia-Migura L, Breslin MF, Davies RH, Woodward MJ. Diversity of strains of Salmonella enterica serotype enteritidis from English poultry farms assessed by multiple genetic fingerprinting. J Clin Microbiol 2001; 39(1): 154-61. [Özet] [Tam Metin] [PDF]
- Swaminathan B, Barrett TJ, Hunter SB, Tauxe RV, CDC PulseNet Task Force. PulseNet: the molecular subtyping network for foodborne bacterial disease surveillance, United States. Emerg Infect Dis 2001; 7(3): 382-9. [Özet] [PDF]
- Kado CI, Liu ST. Rapid procedure for detection and isolation of large and small plasmids. J Bacteriol 1981; 145(3): 1365-73. [Özet] [PDF]
- Hendriksen RS (2003). A global Salmonella surveillance and laboratory support project of the World Health Organization: Laboratory Protocols Level 1 Training Course Identification of Salmonella. 2003, 4th ed. http://www.antimicrobial resistance.dk/data/images/salmonella2-pdf.pdf
- Tenover FC, Arbeit RD, Goering RV, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel electrophoresis: criteria for bacterial strain typing. J Clin Microbiol 1995; 33(9): 2233-9. [PDF]
- Chu C, Wong DW, Wang MH, et al. Genotyping, plasmid analysis, and antimicrobial susceptibility of Salmonella enterica serotype Enteritidis isolates from humans and chickens in central Taiwan. J Formos Med Assoc 2009; 108(10): 765-71. [Özet]
- Gatto AJ, Peters TM, Green J, et al. Distribution of molecular subtypes within Salmonella enterica serotype Enteritidis phage type 4 and S. Typhimurium definitive phage type 104 in nine European countries, 2000-2004: results of an international multi-centre study. Epidemiol Infect 2006; 134(4): 729-36. [Özet] [Tam Metin] [PDF]
- Ward LR, de Sa JD, Rowe B. A phage-typing scheme for Salmonella enteritidis. Epidemiol Infect 1987; 99(2): 291-4. [Özet] [PDF]
- Erdem B, Threlfall EJ, Schofield SL, Ward LR, Rowe B. Plasmid profile typing provides a method for the differentiation of strains of Salmonella enteritidis phage type 4 isolated in Turkey. Lett Appl Microbiol 1994; 19(4): 265-7. [Özet]
- Threlfall EJ, Hampton MD, Ward LR, Rowe B. Application of pulsed-field gel electrophoresis to an international outbreak of Salmonella agona. Emerg Infect Dis 1996; 2(2): 130-2. [PDF]
- Threlfall EJ, Ward LR, Hampton MD, et al. Molecular fingerprinting defines a strain of Salmonella enterica serotype Anatum responsible for an international outbreak associated with formula-dried milk. Epidemiol Infect 1998; 121(2): 289-93. [Özet] [PDF]
- Stanley J, Goldsworthy M, Threlfall EJ. Molecular phylogenetic typing of pandemic isolates of Salmonella enteritidis. FEMS Microbiol Lett 1992; 69(2): 153-60. [Özet]
- Cardinale E, Perrier Gros-Claude JD, Rivoal K, et al. Epidemiological analysis of Salmonella enterica ssp. enterica serovars Hadar, Brancaster and Enteritidis from humans and broiler chickens in Senegal using pulsed-field gel electrophoresis and antibiotic susceptibility. J Appl Microbiol 2005; 99(4): 968-77. [Özet] [Tam Metin] [PDF]
- Aktas Z, Day M, Kayacan CB, Diren S, Threlfall EJ. Molecular characterization of Salmonella Typhimurium and Salmonella Enteritidis by plasmid analysis and pulsed-field gel electrophoresis. Int J Antimicrob Agents 2007; 30(6): 541-5. [Özet]
- Helmuth R, Stephan R, Bunge C, Hoog B, Steinbeck A, Bulling E. Epidemiology of virulence-associated plasmids and outer membrane protein patterns with seven common Salmonella serotypes. Infect Immun 1985; 48(1): 175-82. [Özet] [PDF]
- Lukinmaa S, Schildt R, Rinttila T, Siitonen A. Salmonella Enteritidis phage types 1 and 4: pheno- and genotypic epidemiology of recent outbreaks in Finland. J Clin Microbiol 1999; 37(7): 2176-82. [Özet] [Tam Metin] [PDF]
- Liebana E, Guns D, Garcia-Migura L, Woodward MJ, Clifton-Hadley FA. Molecular typing of Salmonella serotypes prevalent in animals in England: assessment of methodology. J Clin Microbiol 2001; 39(10): 3609-16. [Özet] [Tam Metin] [PDF]
- Liebana E, Clouting C, Garcia-Migura L, et al. Multiple genetic typing of Salmonella Enteritidis phage-types 4, 6, 7, 8 and 13a isolates from animals and humans in the UK. Vet Microbiol 2004; 100(3-4): 189-95. [Özet]
- Fernandez J, Fica A, Ebensperger G, et al. Analysis of molecular epidemiology of Chilean Salmonella enterica serotype enteritidis isolates by pulsed-field gel electrophoresis and bacteriophage typing. J Clin Microbiol 2003; 41(4): 1617-22. [Özet] [Tam Metin] [PDF]
- Avery SM, Liebana E, Hutchison ML, et al. Pulsed field gel electrophoresis of related Escherichia coli O157 isolates associated with beef cattle and comparison with unrelated isolates from animals, meats and humans. Int J Food Microbiol 2004; 92(2): 161-9. [Özet]
- Chung YH, Kwon YI, Kim SY, Kim SH, Lee BK, Chang YH. Antimicrobial susceptibilities and epidemiological analysis of Salmonella enteritidis isolates in Korea by phage typing and pulsed-field gel electrophoresis. J Food Prot 2004; 67(2): 264-70. [Özet]
- De Cesare A, Manfreda G, Dambaugh TR, Guerzoni ME, Franchini A. Automated ribotyping and random amplified polymorphic DNA analysis for molecular typing of Salmonella enteritidis and Salmonella typhimurium strains isolated in Italy. J Appl Microbiol 2001; 91(5): 780-5. [Özet] [Tam Metin] [PDF]
- Selander RK, Beltran P, Smith NH, et al. Evolutionary genetic relationship of clones of Salmonella serovars causing human typhoid and other enteric fevers. Infect Immun 1990; 58(7): 2262-75. [Özet] [PDF]
- Tsen HY, Lin JS. Analysis of Salmonella enteritidis strains isolated from food-poisoning cases in Taiwan by pulsed field gel electrophoresis, plasmid profile and phage typing. J Appl Microbiol 2001; 91(1): 72-9. [Özet] [Tam Metin] [PDF]
- Pang JC, Chiu TH, Chiou CS, et al. Pulsed-field gel electrophoresis, plasmid profiles and phage types for the human isolates of Salmonella enterica serovar Enteritidis obtained over 13 years in Taiwan. J Appl Microbiol 2005; 99(6): 1472-83. [Özet] [Tam Metin] [PDF]
- Pang JC, Chiu TH, Helmuth R, Schroeter A, Guerra B, Tsen HY. A pulsed field gel electrophoresis (PFGE) study that suggest a major world-wide clone of Salmonella enterica serovar Enteritidis. Int J Food Microbiol 2007; 116(3): 305-12. [Özet]
- Murase T, Okitsu T, Suzuki R, et al. Evaluation of DNA fingerprinting by PFGE as an epidemiologic tool for Salmonella infections. Microbiol Immunol 1995; 39(9): 673-6. [Özet]
İletişim (Correspondence):
Dr. Ebru Us,
Ankara Üniversitesi Tıp Fakültesi,
Tıbbi Mikrobiyoloji Anabilim Dalı,
06100 Sıhhiye, Ankara, Türkiye.
Tel (Phone): +90 312 508 3179,
E-posta (E-mail): Ebru.Us@medicine.ankara.edu.tr